Brain tumour segmentation using a triplanar ensemble of U-Nets
基于U网络三平面集成的脑肿瘤分割
Published: May 2021
论文:https://arxiv.org/abs/2105.09511
代码:https://github.com/askerlee/segtran
摘要
脑胶质瘤在其外观和脑MR图像上的位置上都有很大的差异,这使得健壮的肿瘤分割非常具有挑战性,甚至在手工分割中也会导致很高的评分间变异性。在这项工作中,我们提出了一个三平面集成网络,具有一个独立的肿瘤核心预测模块,以准确分割这些肿瘤及其子区域。在MICCAI脑肿瘤分割(BraTS)挑战验证数据集上评估方法,对于肿瘤子区域,获得了增强肿瘤(ET)和肿瘤核心(TC)的Dice相似系数为0.77。在整个肿瘤(WT)区域,获得了0.89的Dice值,这与BraTS的17-19中排名最高的方法相同。我们的方法获得了一个评价分数,在BraTS的20个挑战中,我们的方法排名第10位,在BraTS的20个未公开测试数据集上的ET, WT和TC区域平均Dice值分别为0.81,0.89和0.84。
问题动机:
胶质瘤是最常见的脑肿瘤,具有不同程度的侵袭性,具有高度异质性的亚区域,包括浸润性水肿组织或瘤周水肿(ED)和肿瘤核心区域,包括坏死性核心(NCR)、非增强性肿瘤(NET)和增强性肿瘤(ET)。神经胶质瘤的准确和可重复的自动检测将有助于及时诊断和分期的肿瘤在临床环境,并可靠的分析在大人口研究。然而,神经胶质瘤的内在组织学变异由于MRI扫描中肿瘤特征(例如强度)的异质性而更加复杂。不同的神经胶质瘤亚区域根据其生物条件的不同,在外观和形状上有很大的差异,这使得其分割非常具有挑战性,即使是在专家临床医师跨不同数据集的分割中,也常常导致较高的评分间变异性
思路来源:
MICCAI脑肿瘤分割(BraTS)挑战的目标是在多模态磁共振图像上提供准确的脑肿瘤分割。一些方法,包括最近的深度学习方法,已经在BraTS挑战中被提出。排名最高的方法使用了卷积神经网络(CNN)架构,一些网络主要使用集成网络
[Ensembles of multiple models and architectures for robust brain
tumour segmentation 2017],[Ensembles of densely-connected CNNs with
label-uncertainty for brain tumor segmentation 2018],[Triplanar
Ensemble of 3D-to-2D CNNs with Label-Uncertainty for Brain Tumor
Segmentation 2019]
或编码器-解码器框架。U-Net是最流行的编码器-解码器网络之一,成功地用于肿瘤分割。关于模型维度,2D和3D网络都与额外的后处理步骤一起使用,偶尔也会在多尺度架构和多步级联框架
[Two-Stage Cascaded U-Net: 1st Place Solution to BraTS Challenge 2019
Segmentation Task]、
中使用。一些方法旨在利用二维和三维结构的优势,使用三平面集成CNN
[Triplanar Ensemble of 3D-to-2D CNNs with Label-Uncertainty for Brain
Tumor Segmentation],
比三维网络提供准确的分割和更少的参数。此外,已经提出了对损失函数的改进
[Boundary-aware fully convo-lutional network for brain tumor
segmentation]
以克服类别不平衡,可靠的肿瘤核心检测和准确的肿瘤边界分割。
成果:
作者提出了一种全自动的深度学习方法来分割脑肿瘤,该方法使用一个三平面集成架构,在MR图像的每个平面(轴向、矢状面和冠状面)中包含一个2D U-Net。使用组合损失函数来克服类别不平衡,并包括一个独立的肿瘤核心预测模块来细化肿瘤核心子区域的分割。通过进行消融研究来研究结构的不同组成部分对分割结果的影响。在BraTS20训练和验证数据集上评估了方法,这些数据集在肿瘤特征上表现出广泛的异质性,并为评估分割方法的鲁棒性提供了一个基准。最后,方法在BraTS20测试数据集上的结果表明,作者的方法提供了准确的肿瘤区域分割,排名前10位的最佳方法的挑战。
主要方法:
1.预处理:
将图像裁剪成192×192×160体素的标准尺寸,以便视场(FOV)接近大脑,并对强度值应用高斯归一化。然后分别从192×192、192×160和192×160体素的轴面、矢状面和冠状面体素中提取二维切片。
( 矢状面:矢状面是将人体分切为左右两部分,左右切面就是矢状面,而左右相等的切面被称为正中矢状面。与之相关的名词还有冠状面和横断面。
冠状面:冠状面是沿左,右方向将人体纵切为前后两部分的断面。《人体测量术语》中的定义为:通过铅垂轴与横轴的平面及与其平行的所有平面都称为冠状面。这些平面将人体分成前、后两个部分。
横断面(轴位面):横断面是指过中线桩垂直于中线方向的断面,横断面测量是测量中桩处垂直于中线方向(法线方向)的地面高程,进行横断面测量时首先要测定横断面的方向,然后在这个方向上测定中线桩两侧地面变化点与桩点间的距离和高差,从而绘制横断面图。)
2.CNN架构:
作者使用[Triplanar ensemble U-Net model for white matter hyperintensities segmentation on MR images]中提出的三平面架构。简单地说,如图1所示,三平面结构由三个2D U-Nets组成,每个平面一个,以FLAIR、T1、T1ce和T2片作为输入通道。在训练数据集上,作者观察到,虽然人工分割累积肿瘤核心(TC,即ET + NCR/NET)(图1a)是相当一致的,但由于其潜在的组织学异质性,在单个亚区域(NCR/NET和ET)中存在很大的差异。因此,为了减少ET和NCR/NET标签边界的不一致性,作者修改了ground truth标签,在提供的标签的基础上加入TC,得到ET、ED、NCR/NET和TC 4个标签。在训练过程中,使用ET、NCR/NET和ED标签对三平面结构的U-Net进行培训,使用TC标签对独立的轴向U-Net进行培训,如图1b所示。随后在后处理步骤(参见2.4节)中使用TC区域进一步细化最终输出标签(ET、NCR/NET和ED)。
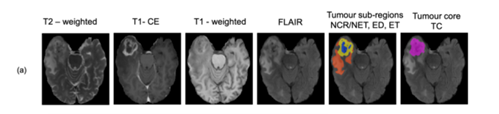
(a)轴向平面的输入模态以及人工分割NCR/NET(蓝色)、ET(黄色)、ED(红色)和TC(洋红)的输入模态
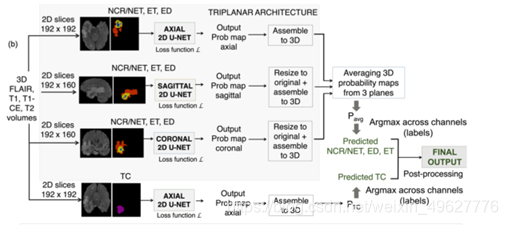
(b)拟议的网络

(c) (b)中使用的3层深U-Net块。为所有U-Net提供4通道(输入模态)的切片
将经典U-Net在每个平面上的深度裁剪为3层(图1b),以减少计算负荷。轴向U-Nets在初始层使用33卷积核,其他两个U-Net使用55卷积核。这有助于了解更多的一般性病变模式,从而避免沿z维分割的任何不连续。在集成模型中,作者使用每个平面上提取的二维切片分别训练每个平面上的U-Net。使用交叉熵(CE)和dice损失(DL)功能的组合,以克服肿瘤/水肿和健康组织之间的类别失衡的影响。损失函数按批计算:
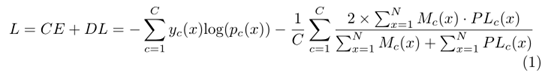
pc(x)表示的输出softmax层,C类标签的数量, Yc (x)∈{0,1}表示每个类在体素x处的二进制值, Mc表示手动分割,PLc表示通过从softmax输出中确定标签的argmax而得到的预测标签映射
在测试过程中,预测作为每个平面上的切片的2D概率图,然后组装成3D体,并调整尺寸至原始尺寸。然后我们将3D概率体平均,得到三平面结构的最终概率体(Pavg)。此外,作者从独立的轴向U-Net中获得了预测TC标签的三维概率图(Ptc)。请注意,3D概率图Pavg和Ptc仍然有一个第4维对应的标签。
3.后处理:
作者通过确定Pavg4维标签的argmax来得到预测的ET、NCR/NET和ED标签图,然后用零填充它们,使它们回到原来的维度。同样,得到了TC在背景下的Ptc作为argmax预测的TC标签图。然后,基于先验知识和人工分割观察到的模式,作者应用了以下附加规则:预测的体积< 200 mm3的ET区域被重新标记为NCR/NET区域的一部分;将TC与ET的差异重新标记为NCR/NET。作者还通过去除ED输出中孤立的杂散噪声区域来进行形态清理(体积< 200mm3,距离最大ED区中心> 75 mm), 并在TC - ED接口中填充缺失的体素,作为ED输出的一部分。
4.数据增强:
数据扩增采用在线方式,从以下变换中随机选择: 平移(x/y偏移∈[- 10,10])、旋转(θ∈[- 10,10])和随机噪声注入(高斯,µ= 0,σ2∈[0.01,0.09]),将所有平面的数据集增加2倍(根据经验选择)。超参数的转换是随机抽样从上述指定的封闭区间使用均匀分布。
5.绩效评价指标:
在线评估平台在BraTS ’ 20中计算的指标如下:
(i)dice相似系数为2×TP/(2×TP+FP+FN)
(ii)以TP/(TP+FN)测量的灵敏度
(iii)以TN/(TN+FP)测量的特异性
(iv) Hausdorff距离的95%百分位数(H95)
其中TP、FP、FN、TN分别为真阳性、假阳性、假阴性、真阴性体素数。
对于真实标签,手工分割包括ET、ED和NCR/NET类别,评估平台对以下肿瘤分区确定评价绩效指标:
(1) ET,(2) TC (NCR/NET+ET)和(3)整个肿瘤(WT, TC+ED)
实验与结果:
标记训练数据的交叉验证:使用来自BraTS 20个训练数据的369名标记受试者进行5折交叉验证,训练-验证-测试的分裂比为255-37-73人(最后一折为255-37-77人)。测试分割(使用挑战在线平台评估)的结果如表1所示。在三个子区域中,对整个肿瘤(WT)的分割性能最好,Dice的平均值为0.93。图2显示了我们的方法的一些样本输出以及手工分割。
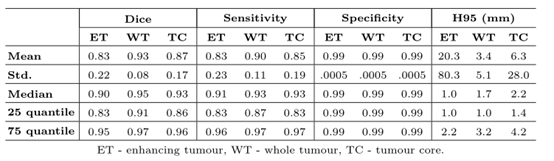
表1:BraTS的20个训练数据的5倍交叉验证结果。
标记训练数据的消融实验:
为了确定架构中的各个组件对分割性能的影响,作者使用以下组件评估分割结果:
(i)仅轴向2D U-Net,
(ii)轴向+矢状2D U-Net,
(iii)三面网(轴向+矢状+冠状U-Net)
(iv)三面网+轴向U-Net用于TC标签检测。
作者使用了交叉验证策略(与上面提到的训练验证-测试分割相同)来评估性能指标。消融实验的性能指标值如表2,其箱线图如图3所示。随着TC网络的加入,子区域的分割得到改善,Dice和敏感性值显著增加(p < 0.01),尤其是ET和TC子区域。作者还观察到,与单独的U-Nets相比,使用三平面结构的WT分割的特异性有显著改善(p<0.001)。

对两个样本受试者(顶部和底部)的20个BraTS训练数据的结果。对两个样本受试者(NCR/NET -蓝色,ET -黄色和ED -红色)的轴向切片进行手动分割和预测输出。(下面ET=0 在底部的面板中,白色箭头表示对ET区域的错误预测,导致Dice值为0.00。)
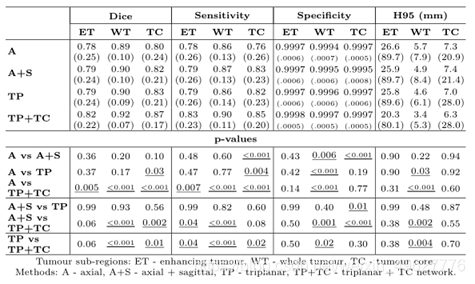
表2:BraTS 20个训练数据消融实验结果。肿瘤子区域报告的方法中不同成分的平均值和标准差(括号内)值。
对未标记验证数据的评价:
将训练数据的5折交叉验证训练出的模型应用于验证数据,最终预测采用多数投票(来自5个模型)。在线评价平台的结果如表3所示。图4显示了方法的一些样本验证数据输出和输入模式。结果与训练数据相似,WT分割结果最好平均Dice值为0.89,H95值为4.4。
测试数据结果:
最后,在线评价平台对BraTS的20个测试数据的结果如表4所示。与验证阶段相似,对5个模型(来自5折交叉验证)的结果进行多数投票来预测肿瘤区域标签。从表中可以看出,在验证阶段,WT区域的Dice值高于ET和TC区域。然而,ET和TC区域的Dice和敏感性值明显高于验证数据,几乎与训练数据集上的重复验证结果相同。
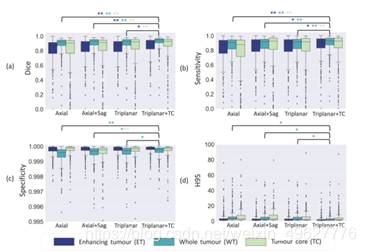
(a)Dice,(b)敏感性,©特异性和(d)轴向,轴向+矢状面,三平面和三平面+TC病例的H95。对于肿瘤子区域,表现指标的显著差异用相应颜色的星号表示(* - p<0.01,**-p<0.001)。请注意,只显示了三平面+TC和其他情况之间的性能指标的显著差异(对于单个对的所有组合,请参见表2)。
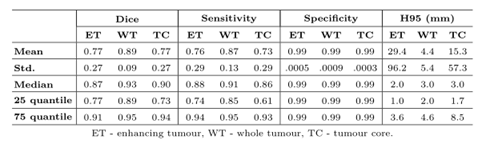
表3 BraTS 20验证数据的结果。
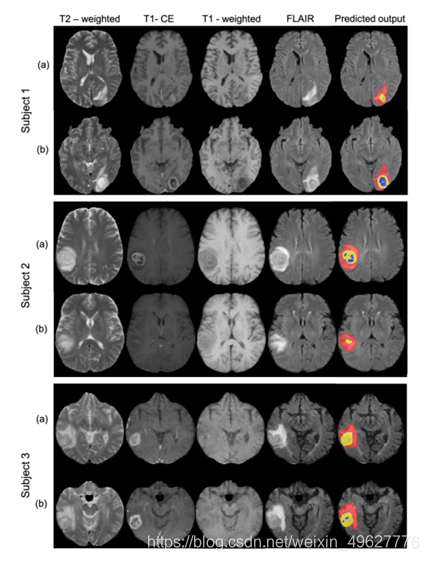
BraTS20验证数据中三个样本受试者。三个样本受试者(NCR/NET -蓝色,ET -黄色和ED -红色)的两个轴向切片(a)和(b)的预测输出
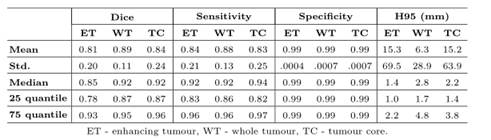
表4:BraTS20测试数据的结果
总结:
在这项工作中,作者提出了一种端到端自动肿瘤分割方法,使用二维U-Net的三平面集成架构。作者的方法在训练数据集上以0.83,0.93和0.87的dice值分割肿瘤的ET, WT和TC子区域。在一个独立的无标记验证数据集上,方法获得了ET, WT和TC子区域分割的Dice值为0.77,0.89和0.77。在BraTS20测试数据集上,方法获得了ET、WT和TC区域的Dice值分别为0.81、0.89和0.84。
研究单个成分对分割性能的影响有助于更好地理解所提出的方法。对于所有的组织类别,单一轴向网络表现最差,可能是由于缺乏来自连续切片的上下文信息。三平面结构提供了比单个二维网络更好的性能,具有更高的dice和灵敏度值。此外,通过添加特定的TC网络,特别是对于TC类,实现了性能指标的显著改进。WT的分割随着每一个成分的加入而改善,显著降低的H95值表明肿瘤的分割更加精确。
从训练和验证数据的结果来看,WT的表现高于其他两类,这表明该方法分割累积肿瘤区域(包括水肿/侵犯区域),比区分核心和水肿具有更高的准确性。肿瘤分类错误然而,几个核心地区是由于情况等类是非常小的(白色箭头在图2中所显示的那样)。这导致ET被错误地预测(通过网络)或错误重新标记(在后处理步骤)作为NCR /网络类的一部分。这在很大程度上影响了该科目ET班级的骰子分数。总的来说,观察到,由于肿瘤特征的广泛范围和真实标签的变化,对TC和ET类应用一般化/一致的先验信息或后处理操作是不可能的,这对分割任务提出了一个主要挑战。有趣的是,对于TC区域,与验证数据集相比,作者的方法在训练和未见测试数据集上表现得更好,从表1和4中的指标的较高值观察到,与表3中的相比。训练数据集上的值较高可能是因为交叉验证结果通常更容易过拟合,因此与未见验证数据上的结果相比,其可靠性较低。然而,作者的方法在不可见的测试数据集上也提供了一贯良好的性能(表4)。由于这是一个不可见的数据集,不能排除测试数据集的肿瘤特征和图像强度轮廓可能更类似于训练数据集的可能性。
综上所述,提出的三平面集成方法从BraTS 20挑战的多模态数据中实现了对整个肿瘤及其子区域的准确分割。在挑战之后,将公开方法作为Docker容器,可以用作一个独立的肿瘤分割工具。未来的研究方向包括利用显著特征(例如使用注意网络)进一步改善肿瘤分区分割。