摘要
肿瘤的高通量基因组和分子分析正在成为重要的临床方法,越来越多的分子分析被用于指导癌症患者的护理,特别是在晚期和不可治愈的癌症中。然而,浏览科学文献去进行基于分子普分析的临床决定对许多肿瘤临床医生和研究人员来说都是耗时耗力的。个性化癌症治疗网站(www.personalizedcancertherapy.org)的建立为临床医生和研究人员提供一个在线资源,以促进可用数据的导航。具体来说,这个资源可以用来帮助致癌基因改变的患者确定潜在的治疗方案。
1 网站基因信息
单个基因表是网站布局的基础。为了充分注释并在网站上公布,基因必须满足一系列初始可行性标准:(i)基因必须是与癌症发展或进展相关;(ii)必须有证据表明在治疗上靶向基因或其途径可能导致肿瘤抑制;(iii)靶向基因产物或其下游结果(例如下游信号传导)的药物是FDA批准的或正在进行临床研究.PODS小组的成员基于现有可用科学数据的强度选择基因,同时也从MD Anderson临床医生和研究人员的输入中选择基因。一旦选择了一个基因进行注释,系统的科学文献回顾就会生成网站基因页面部分的内容(图1)。大部分内容是使用PubMed或互联网搜索引擎的手工文献综述生成的,但网站也在开发和使用半自动化的方法。

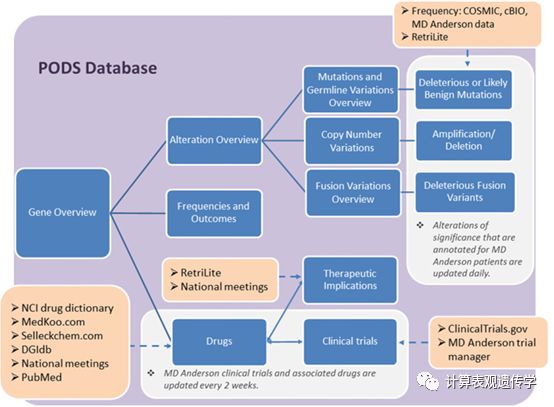
1 单核苷酸或基因融合变体的功能注释
对于变体的功能注释,我们在PODS数据库中编译独特的变更。这包括体细胞或种系单核苷酸改变,以及小的插入或缺失。用于功能注释的资源包括cBIO,COSMIC,下一代测序平台中包括的改变,在个别病人报告的注释过程中遇到的更改,以及发表文章中的发现。
网站手动审查检索的文献并编写变体的功能注释。每个变体的优先级都根据其在咨询数据库中报告的频率,公布的功能,或治疗的数据确定。只有明确的临床或临床前数据的变化才会发布到网站上。
1 药物注释:确定药物-基因关联
为了生成与感兴趣基因相关的药物列表,网站使用在线数据库和科学文献对药物进行分类。这些数据库包括美国国家癌症研究所(NCI)药物词典,Selleckchem.com,medkoo.com,药物基因相互作用数据库(DGIdb)和ClinicalTrials.gov。
然后,网站标记每种药物与潜在的相关生物标志物。 目前对于PODS,生物标志物是指基因组或蛋白质表达水平的改变。网站记录的报道从每个变更背景下的科学文献的特定药物的IC50,然后根据专一性对每个直接和间接药物靶标进行排序。如果药物在生化检测中能够抑制基因编码蛋白的活性(IC50 <1 mmol / L),则认为基因是药物的直接靶点;如果药物靶向的蛋白质位于感兴趣的基因编码的蛋白质的途径内,则基因被认为是药物的间接靶标。另外,当存在感兴趣基因内的改变时,必须有证据表明该药具有抗肿瘤特性。
FDA批准的药物也标有每个批准的疾病类型,生物标志物和任何其他可联合使用的药物。只有直接或间接定位感兴趣基因的临床上可用的药物(即经FDA批准或正在临床试验中进行积极调查的药物)才会自动发布到各自基因页面的“药物”选项卡上。
1 治疗影响
为了描述携带特定生物标志的特定肿瘤类型中,特定药物的有效性水平,我们对治疗效果进行注释。RetriLite用于从MEDLINE摘要中检索与潜在治疗含义相关的科学文献。然后对文献进行手动评估,并对药物,基因改变,疾病和证据水平进行分层。
分层级别如下:级别1:包括具有与生物标志物评估(在特定疾病的情况下)相关的治疗功效的强有力证据的前瞻性研究,通常在生物标志物的情况下经FDA批准该药物; 级别2:包括对特定肿瘤类型的回顾性研究或来自另一种肿瘤类型的临床数据; 级别3:包括具有科学依据的案例研究或将生物标志物与药物功效联系起来的高质量临床前数据。网站还报告了相互矛盾的数据,以及生物标志物可能赋予对特定药物或某类药物的抗性的证据。
1 临床试验注释
我们使用内部信息学工具来确定临床试验。 试验检索利用PODS数据库中注释药物的直接和间接基因目标信息。 一旦为感兴趣的基因产生了一系列试验,每个试验都通过汇总来自Clinical- Trials.gov和MD Anderson的内部临床试验管理系统的信息加以注释.
当一个基因进入系统时,使用之前在数据库中注释的与该基因相关的药物进行试验。临床试验信息学工具以编程方式与ClinicalTrials.gov的Web应用程序编程接口(API)进行交互。通过这个,我们可以检索使用一种或多种有针对性的感兴趣的疗法治疗癌症患者的公开试验。 PODS小组随后手动注释这些试验的药物,疾病和生物标志物。试验被指定为“基因型选择”(即,需要特定基因组改变用于登记的试验)或“基因型相关的”。
参考文献:
Kurnit, K.C., et al., "PersonalizedCancer Therapy": A Publicly Available Precision Oncology Resource.Cancer Res, 2017. 77(21): p.e123-e126.
往期「精彩内容」,点击回顾
DNA测序历史 | CircRNA数据库 | Epigenie表观综合 | 癌症定位
BWA介绍 | 源码安装R包 | CancerLocator | lme4 | 450K分析
乳腺癌异质性 | BS-Seq | 隐马模型 | Circos安装 | Circos画图
KEGG标记基因 | GDSC | Meta分析 | R线性回归和相关矩阵
精彩会议及课程,点击回顾