当我们处理高维数据时,很难直观地理解和发现数据中的结构和关联。t-SNE是一种强大的降维技术,能够揭示高维数据背后的秘密。本文将主要介绍t-SNE的原理和应用。
一、t-SNE的核心思想
t-SNE(t-Distributed Stochastic Neighbor Embedding)是一种用于高维数据可视化和降维的强大技术。这个算法是由Maaten和Hinton在2008年首次提出的[1]。
在提出t-SNE之前,已经有一些降维和可视化技术,如PCA(主成分分析)和LLE(局部线性嵌入)。然而,这些方法在处理高维非线性数据时存在局限性。为了克服这些局限性,t-SNE算法应运而生,旨在更好地保留高维数据的局部结构。
t-SNE算法的主要贡献在于它使用了一种基于概率的方法来测量高维数据点之间的相似度,并在低维空间中尽量保持这些相似度。t-SNE使用了一个特殊的概率分布(t分布),能够有效地处理高维数据中的异常值,并在低维空间中生成更好的聚类效果。
自从t-SNE算法被提出以来,它已经广泛应用于各种领域,如生物信息学、图像处理、自然语言处理和社交网络分析等。许多研究人员和工程师都发现t-SNE是一种非常有效的方法,能够揭示高维数据中隐藏的结构和关联。

维度复杂的世界
二、t-SNE的原理是什么?
t-SNE的工作原理分为以下几个步骤:
- 计算高维空间中数据点之间的相似度。t-SNE会为每个数据点计算一个概率分布,表示它与其他数据点的相似度。这个概率分布可以看作是一种“邻居关系”。
- 在低维空间中为每个数据点找到一个新的位置,并计算低维空间中数据点之间的相似度。t-SNE同样会为低维空间中的每个数据点计算一个概率分布。
- 最小化高维空间和低维空间中概率分布之间的差异。t-SNE采用一种名为KL散度(Kullback-Leibler Divergence)的优化方法来衡量这两个概率分布之间的差异,并通过梯度下降等算法来最小化这个差异。通过这种方式,t-SNE可以使得低维空间中的数据点分布尽量保持高维空间中的相似关系。
值得注意的是,t-SNE中的“t-分布”是一种特殊的概率分布函数,它在低维空间中有利于保留局部结构,同时对于远离的数据点,它会赋予较大的权重。这有助于在低维空间中更好地展示数据的结构。
三、为什么用t-SNE?
t-SNE和KPCA都是非线性降维方法,它们在处理高维数据时具有各自的优缺点:
| t-SNE的优点 | t-SNE的缺点 |
|---|---|
| 保留局部结构:t-SNE非常擅长保留高维数据中的局部结构,在降维后的低维空间中,相似的数据点会聚集在一起,形成明显的类簇。 | 计算复杂度高:t-SNE的计算复杂度较高,对于大规模数据集的降维可能需要较长的计算时间。 |
| 可视化效果好:t-SNE在可视化高维数据时的效果很好,尤其是在展示复杂数据集时,它能清晰地显示不同类别和簇之间的关系。 | 难以选择最优参数:t-SNE有一些需要调整的参数,如学习率和感知半径,这些参数的选择可能会影响降维结果。 |
四、t-SNE的几个重要参数
1.放大系数(Exaggeration)
用于设置数据中自然簇的大小,在t-SNE算法的初始阶段,放大系数用于放大高维空间中的相似度概率。这有助于更好地分离数据点,形成清晰的类簇。在实操中,这个系数有时候并不会对聚类效果的改善有很大提升,所以大家酌情赋值吧。该参数需不小于1,在MATLAB中放大系数的默认值为4。
2.困惑度(Perplexity)
这是一个衡量高维空间中数据点邻居数量的参数。困惑度越大,算法考虑的局部邻居数量越多,这将影响到局部结构的保留。合适的困惑度值取决于数据集的大小和复杂性。MATLAB帮助中表示,困惑度值的范围在5到50之间,但是该数值还是要结合具体数据集的大小来设定,说白了它就是预估每个聚类可能有多少个元素。MATLAB中该值默认为30。
3.学习率(LearnRate)
t-SNE使用梯度下降来优化低维空间的数据点位置。学习率决定了梯度下降的步长。如果学习率过大,算法可能无法收敛,导致降维结果不稳定;如果学习率过小,算法可能会陷入局部最优解,收敛速度变慢。通常,学习率的值可以设置在10到1000之间。
4.降维后的维度(NumDimensions)
该参数是需要同学们自己指定的,在实际使用中通常需要结合实际应用场景进行设置。
需要注意的是与PCA、KPCA等降维方法不同,t-SNE使用了随机梯度下降算法优化低维空间的数据点布局,它没有明确地计算协方差矩阵和特征值。所以也就不能像PCA和KPCA方法一样通过贡献度比率进行维度设置。
下边我们举例说明一下。
五、案例:降维、聚类与分类
举一个PCA中介绍过的例子。
这里介绍一下鸢尾花数据集,鸢尾花在机器学习里是常客之一。数据集由具有150个实例组成,其特征数据包括四个:萼片长、萼片宽、花瓣长、花瓣宽。数据集中一共包括三种鸢尾花,分别叫做Setosa、Versicolor、Virginica,就像下图:

也就是说这组数据的维度是150*4,数据是有标签的。(有标签是指每个实例我们都知道它对应的类别)
下边我们看一下不同参数设置对聚类效果的影响:
1.首先是放大系数(Exaggeration)
我们将Exaggeration设置成1,数据降维成2维后,分布图如下:
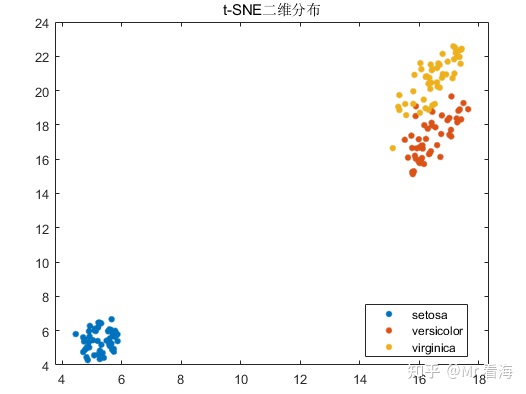
此时再将Exaggeration设置为比较夸张的大值,比如20,此时分布图如下:
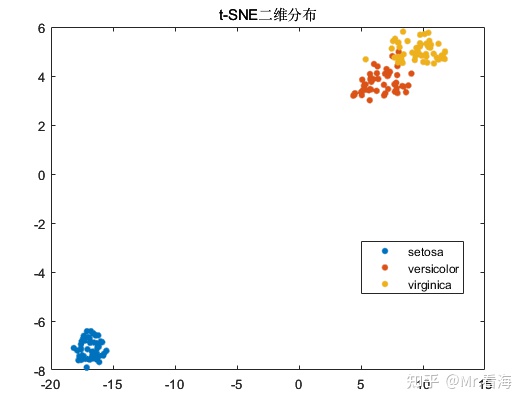
是的,变化并不算明显。
2.困惑度(Perplexity)
Perplexity就是预估每个聚类可能有多少个元素,在这个例子中,每个聚类中包含了50个点,所以Perplexity最合适的赋值就是50,此时画出来的图像如下(Exaggeration设置成了默认值4):
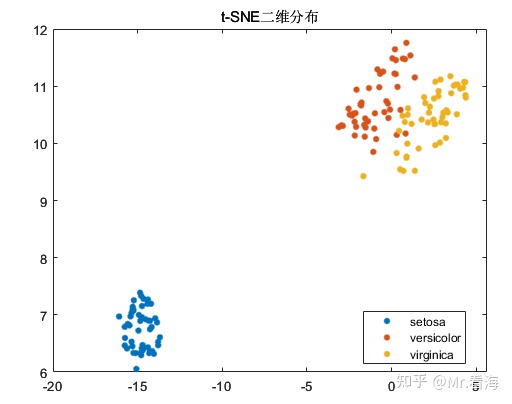
如果把Perplexity改的明显偏小(比如5),就会变成下图这样:

如果把Perplexity改的明显偏大(比如100),则会变成:
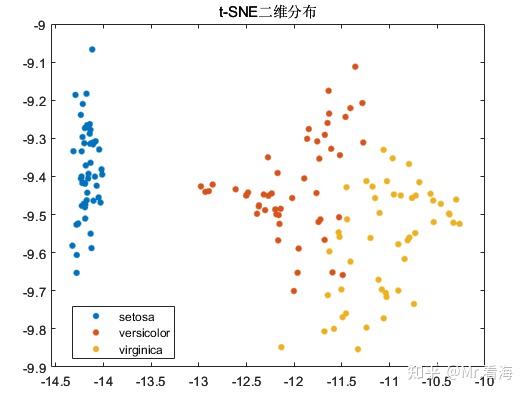
可见,选择合适的 perplexity 值对于获得良好的降维和可视化效果至关重要。
3.学习率(LearnRate)
学习率主要影响收敛速度,学习率过小,可能导致算法收敛速度变慢,需要更多的迭代次数才能找到一个较好的低维表示;学习率过大,可能导致算法在优化过程中发散,无法收敛到一个稳定的低维表示。t-SNE 算法的结果可能因初始化和随机性而具有一定程度的不稳定性。为了获得更稳定的结果,可以多次运行 t-SNE 算法,并比较不同学习率下的降维效果。
LearnRate设置成100时:
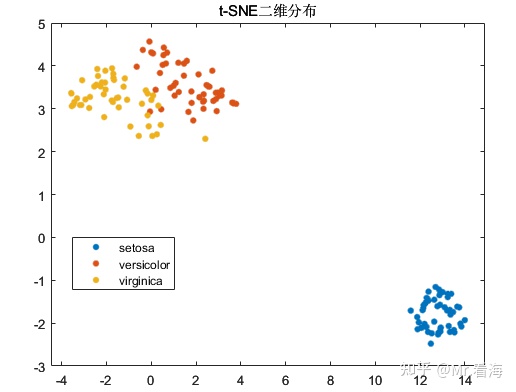
LearnRate设置成1000时:
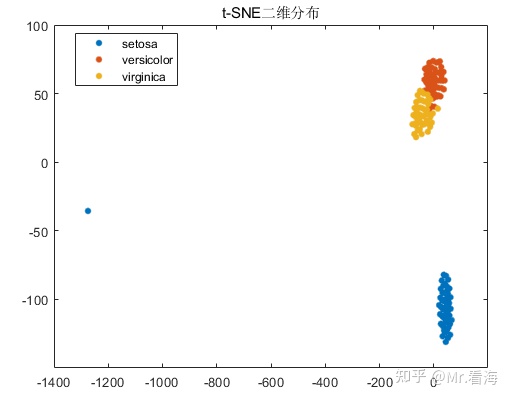
4.降维后的维度(NumDimensions)
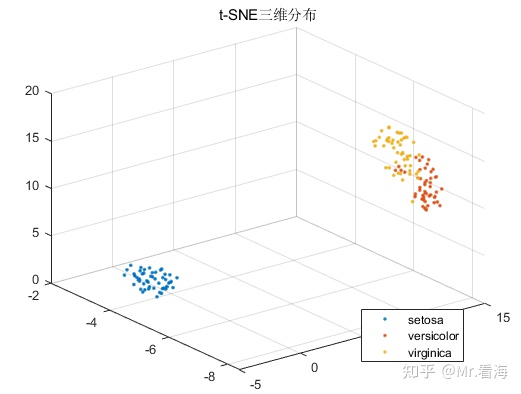
当NumDimensions设置为2时,绘制的图片是上述这样的二维图;当NumDimensions设置为3时,则可以绘制三维图像,当然也可以将高位数据降维为4维及以上维度,但是就无法画图了。当NumDimensions为3时,绘制出的三维图就会像这样(此时Exaggeration=4、Perplexity=50、LearnRate=500):

尽管t-SNE算法的初衷是降维而非聚类,不过由于t-SNE降维后的数据常常会用做机器学习的输入数据,在数据降维的同时查看降维后数据的分布情况,对于模式识别/分类任务的中间状态确定还是十分有益的,再直白些说,这些图片放在论文里丰富一下内容也是极好的。
在这种应用场景下,数据降维的最主要目的其实还是解决数据特征过于庞大的问题,这个例子中特征只有4个,所以还不太明显。很多时候我们面对的是几十上百乃至更多的特征维度,这些特征中包含着大量冗余信息,使得计算任务变得非常繁重,调参的难度和会大大增加。此时加入一步数据降维就是十分有必要的了。
六、MATLAB的t-SNE降维快速实现
t-SNE算法在MATLAB中有官方函数,名字就叫做tsne,熟悉编程的同学可以直接调用。
对于不熟悉MATLAB编程,或者希望更简洁的方法实现t-SNE降维,并实现可视化,则可以考虑使用本专栏封装的函数,它可以实现:
1.输入数据的行列方向纠正。是的,MATLAB的pca函数对特征矩阵的输入方向是有要求的,如果搞不清,程序可以帮你自动纠正。
options.autoDir = 'on'; %是否进行自动纠错,'on'为是,否则为否。开启自动纠错后会智能调整数据的行列方向。2.指定输出的维度。也就是降维之后的维度,当然这个数不能大于输入数据的特征维度。
options.NumDimensions = 3; %降维后的数据维度3.数据归一化。你可以选择在PCA之前,对特征数据进行归一化,这也只需要设置一个参数。
options.Standardize = false; %输入数据是否进行标准化,false (默认) | true 4.绘制特征分布图。在降维维度为2或者3时,可以绘制特征分布图,当然你也可以选择设置不画图,图个清静。
figflag = 'on'; %是否画图,'on'为画图,'off'为不画,只有NumDimensions为2或者3时起作用,3以上无法画图5.设置其他与t-SNE相关的参数。包括我们上边提到的Exaggeration、Perplexity、LearnRate等参数。
options.Exaggeration = 4; %数据中自然聚类的大小,整型数据
options.Perplexity = 50; %即每个点的有效局部邻居数
options.LearnRate = 500; %优化过程的学习速率,指定为正标量。通常设置值为100到1000。设置好这些配置参数后,只需要调用下边这行代码:
rsneFea = khKPCA(data,options,species,figflag);%rsneFea为降维后的数据矩阵就可以绘制出这样一张图:
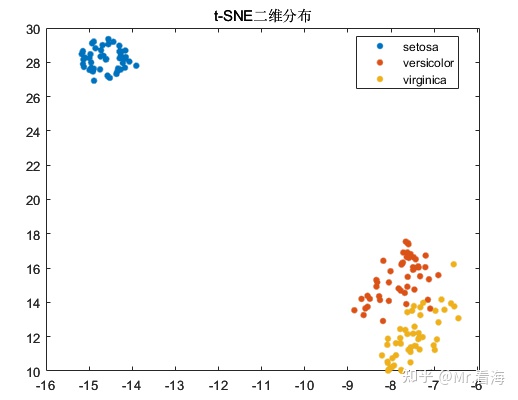
如果要绘制二维图,把options.NumDimensions设置成2就好了。绘制出来是这样:
不过上述是知道标签值species的情况,如果不知道标签值,设置species=[]就行了,此时画出来的分布图是单一颜色的。
上述代码秉承了本专栏一向的易用属性,功能全部集中在kTSNE函数里了,这个函数更详细的介绍如下:
function tsneVal = kTSNE(Fea,options,species,figflag)
%% 执行数据的t-sne降维,需要MATLAB2017a及以上版本
%% 可以实现2维、3维以及更高维度的降维,只有二维和三维可以画图
% 输入:
% Fea:待降维数据,R*Q的矩阵,R为批次数,Q为特征维度,例如特征维度为8的共100组数,tempFea的维度应为100*8。输入该变量时一定要注意行列方向是否正确,如不正确需要转置
% options:一些与tsne降维有关的设置,使用结构体方式赋值,比如 options.Algorithm = 'barneshut',具体包括:
% -Algorithm:tsne算法,可选:'barneshut' (默认) |'exact'。其中'barneshut'执行近似优化,数据量大时,速度更快。
% -Distance:距离量度方法选择,可选:'euclidean' (default) | 'seuclidean' | 'cityblock' | 'chebychev' | 'minkowski' | 'mahalanobis' | 'cosine' | 'correlation' | 'spearman' | 'hamming' | 'jaccard'
% 具体描述见:https://ww2.mathworks.cn/help/stats/tsne.html?s_tid=doc_ta
% -Exaggeration:数据中自然聚类的大小,整型数据,默认为4
% -NumDimensions:降维后的数据维度,默认为2
% -LearnRate:优化过程的学习速率,指定为正标量。通常设置值为100到1000。
% -Standardize:输入数据是否进行标准化,false (默认) | true
% -Perplexity:困惑度。即每个点的有效局部邻居数,指定为正标量。更大的困惑度会使更多的点作为最近的邻居。一般情况下,困惑度值的范围在5到50之间
% species:分组变量,可以是数组、数值向量、字符数组、字符串数组等,但是需要注意此变量维度需要与Fea的组数一致。该变量可以不赋值,调用时对应位置写为[]即可
% 例如species可以是[1,1,1,2,2,2,3,3,3]这样的数组,代表了Fea前3行数据为第1组,4-6行数据为第2组,7-9行数据为第三组。
% 关于此species变量更多信息,可以查看下述链接中的"Grouping variable":
% https://ww2.mathworks.cn/help/stats/gscatter.html?s_tid=doc_ta#d124e492252
%
% figflag:是否画图,'on'为画图,'off'为不画,只有NumDimensions为2或者3时起作用,3以上无法画图
需要上边这个函数文件以及测试代码的同学,可以在下边链接获取:
参考
- ^Maaten L,Hinton G. Visualizing data using t-SNE[J].Journal of machine learning research,2008,9:2579-2605.