目录
摘要
方法
框架
landmark感知对齐
平面特定图集构建
测试体积图集对齐
自适应动态终止
实验
Dataset Setting
参数
RL
RNN(自适应动态终止Adaptive Dynamic Termination)
3D U-net(检测胎儿脑landmarks)
评定标准
结果
与最先进的方法进行比较
地标标齐模块的影响
自适应动态终止的分析
显著差异分析
来自SP的临床生物特征评估
定性评价
博文相关链接
论文References
| 具有热启动和自适应动态终止的智能体,用于 3D 超声中的平面定位[TMI 2021] [官方代码] |
智能体声压级AgentSPL |
特征点对齐、强化学习、CNN、RNN、SP 定位 |
摘要
在SP定位中,agent可能无法捕获目标SP,并在没有终止条件的情况下继续探索。
可使用进一步的终止动作[6]来扩展动作空间。但是,扩大行动空间会导致训练不足。
一些工作通过检测振荡[7]或最低q值[9]来终止代理搜索。虽然没有引入额外的操作,但这些方法仍然需要代理以最大的步骤完成推理,这是低效的。
agent搜索过程中,经常被困在局部最小值中,因此很难估计最优终止步骤。
因此,在SP定位任务中,采用动态终止策略来确保SP定位的有效性和效率是非常可取的。
新设计的自适应动态终止来增强之前的RL框架,使代理搜索能够提前停止,最多节省67%的推理时间,从而同时提高了RL框架的准确性和效率。
方法
(本文只讲改善处,未改处见之前的RL框架)
框架

图2 框架
框架包含三个模块:
1)landmark感知对齐,为高效的agent搜索提供了warm start(左)
2)基于深度RL的SP定位(中间)
3)基于学习的自适应终止,以提高定位效率和准确性(右)。
landmark感知对齐

图4 landmark感知对齐模块的Pipeline
[8]中提出了一个landmark感知对齐模块,作为通过解剖先验知识的搜索过程的专用warm start。
在本节中详细介绍了一个更具体的处理管道。这个landmark意识模块将US的体积与地图集空间对齐,从而减少了胎儿姿势和超声采集的多样性。
如图4所示,提出的对齐模块包括两个步骤,即平面特定的图谱构建、测试体积-图谱对齐。具体情况描述如下。

Θ计算平面法向量之间的夹角,参照第三节-C节中的公式5。
平面特定图集构建
在本研究中,构建图集,通过基于landmark的 配准初始化 测试体中的SP定位。
因此,从训练数据集中选择的landmark集需要同时含用于注册的标志、用于平面初始化的SP参数。如图4所示,建议为每个SP选择特定的图谱,以提高定位精度。而不是每个SP[3]选择一个共同的解剖模型
[3]Diagnostic Plane Extraction from 3DParametric Surface of the Fetal Cranium胎儿颅骨三维参数表面的诊断平面提取_luemeon的博客-CSDN博客
为了确保初始化的有效性,理想情况下,所选地图集的特定SP应尽可能接近其他训练体积的SPs。
算法1显示了基于最小平面误差(即两个平面之间的角度和距离之和)从训练数据集中确定特定平面的图谱体积。
在训练阶段,首先将每个体积作为一个初始化的agent图集,然后对剩余的体积进行基于landmark的刚性配准。
根据每个agent图集的线性注册平面与标准平面真实值之间测量的平均平面误差,选择误差最小的体积作为最终图集。
测试体积图集对齐
对齐模块是基于landmark检测和匹配,与直接回归不同。
将landmark检测转换为热图回归任务[37],以避免学习高度抽象的映射函数(即特征表示为landmark坐标)。用l2-范数回归损失训练了一个定制的3D U-net [38],表示为:

其中N = 3为landmark数,Hi、ˆHi分别表示第i个预测的landmark热图和标准landmark热图。
这些标准landmark热图是通过在相应的地标位置放置一个高斯核而创建的。
在推理过程中,将测试量传递给landmark探测器,以得到预测的landmark热图。
选择landmark热图中值最大的坐标作为最终预测。
通过由landmark计算的变换矩阵将体积映射到地图集空间,从而为agent创建一个有界的环境。
此外,利用图集的标注目标平面函数作为agent的初始起始平面函数。
自适应动态终止
考虑到迭代交互作用的顺序特征,如图2所示,用一个附加的RNN模型对q值序列与最优步长之间的映射进行建模。
Q值定义为qt = {q1,qi,...,q8},在迭代t时由8个候选动作组成;
Q值序列为时间序列矩阵Q = [q1,q2,...,qn],其中n表示迭代步骤的索引。
以q值序列为输入,RNN模型可以基于最高角度和距离改进(ADI)学习最优终止步骤。
(ADI的定义参照III-C节中的公式7。)
在训练过程中,我们从q值序列中随机抽取子序列作为训练数据,并将采样区间内最高的ADI表示为标准切面。
与之前的研究[8],[9]不同,我们设计了一种动态终止策略来提高强化框架的推理效率。
具体来说,我们的RNN模型基于当前的零填充q值序列,每两次迭代执行一次推断,
从而允许在迭代步骤中提前停止,并有前三个重复的预测。
之前的研究[8]使用平均绝对误差(MAE)损失函数来训练终止模块中的RNN。
然而,它具有恒定的反向传播梯度,且缺乏测量细粒度误差。
本研究用均方误差(MSE)损失函数代替它,以重温这一点,并针对一个更稳定的训练过程。
由于标准切面,即最优终止步长,通常大于1(例如10∼75),传统的MSE损失函数在训练中可能由于梯度过多而难以收敛。
采用具有平衡超参数的MSE损失函数,定义为
其中,w为RNN参数,x为RNN的输入序列,f(x;w)表示RNN网络,G表示最优终止步骤。
平衡超参数δ = 0.01可以将学习到的步骤的值范围近似归一化为[0,0.75],从而简化了训练过程。RNN模型使用从训练量中得到的推理结果进行训练。
实验
Dataset Setting
使用三个不同的三维US数据集验证了所提出的框架,
包括胎儿大脑、胎儿腹部、子宫。
具体来说,目标是定位三个SPs:
经脑室(TV)、经丘脑(TT)和经小脑(TC)、
胎儿腹部(AM)SP、
子宫中矢状面(S)、横向(T)和冠状面(C) SPs。
从每个胎儿/子宫US体积中选择3/4个标志:胼胝体膝和压部,小脑蚓部中心作为胎儿脑容量;
脐静脉入口、中心、胆囊颈部为胎儿腹部体积;
两个子宫内膜子宫角,子宫内膜子宫底部和子宫壁底部为子宫体积。
收集了1635个产前3D US体积的数据集
433个胎儿大脑,519个胎儿腹部和683个子宫US体积
随机分割数据集,分别进行训练、验证、测试
胎儿脑313、20、100,胎儿腹部389、20、110,子宫519、20、144
数据集的平均超声体积大小为胎儿为270×207×235,子宫为261×175×277
所有的图像都被重新采样到各向同性的体素大小,统一0.5×0.5×0.5mm3
(各向同性就是各个方向体素相同,如文件的体素空间(spacing)为 1mm*1mm*1mm
各向异性就是各个方向体素不同, 如文件的体素空间(spacing)为 1mm*1mm*5mm)
4名有5年经验的超声技师为所有超声体积提供了特征点landmark和标准切面sp的手动注释。
所有注释结果均在一位有20年工作经验的资深专家的严格质量控制下进行了复核。
经当地机构审查委员会批准,所有超声都是匿名的,并由专家使用带有集成3D探头的迈瑞DC-9超声系统获得。
- train是训练集,
- val是训练过程中的测试集,是为了在边训练边看到训练的结果,及时判断学习状态。来验证是否过拟合、以及用来调节训练参数等。与train没有交集,对最终训练出的模型没有贡献。
- test就是训练模型结束后,用于评价模型结果的测试集。
只有train就可以训练,val不是必须的,比例也可以设置很小。
test对于model训练也不是必须的,但是一般都要预留一些用来检测,通常推荐比例是8:1:1
但现在很多模型都不用validation了。
现在模型中防止过拟合的机制已经比较完善了,Dropout\BN等做的很好了。
而且很多时候都用原来的模型进行fine tune,也比从头开始更难过拟合。
所以大家一般都定一个训练迭代次数,直接取最后的模型来测试。
参数
RL
Adam优化器[40]对整个框架进行了训练,
对DDQN进行了 epoch =100(约4天)的训练、
将损失函数(公式2)中的discount factor(折扣因子) γ设为0.9。(γ∈[0,1])
目标Q network每1500次迭代复制一次Q network的参数。
胎儿数据集的最大迭代次数为75次,子宫数据集中为30次,为药物探索保留足够的移动空间。
 −选择策略[17]的初始首先设置为0.6,
−选择策略[17]的初始首先设置为0.6,
在训练期间每10000次迭代乘以1.01,直到0.95。被训练了100个epochs,
# =============== define training
# batch size, INT
self.batch_size = 4
# target net weight update term, INT
# 目标Q network每1500次迭代复制一次当前Q network的参数
self.target_step_counter = 1500
# learning rate, FLOAT
self.lr = 5e-5
# weight decay, FLOAT
self.weight_decay = 1e-4
# reward decay γ, FLOAT
self.gamma = 0.95
# memory capacity, INT 有优先级的Replay-buffffer
self.memory_capacity = 15000
# epsilon for the greedy, FLOAT
self.epsilon = 0.6
# =============== define default
# gpu id
self.gpu_id = 0
# total epoch
self.num_epoch = 100
# max steps(default fetal,uterus子宫30)
self.max_step = 75
RNN(自适应动态终止Adaptive Dynamic Termination)
采用具有平衡超参数的MSE损失函数,定义为

其中,w为RNN参数,x为RNN的输入序列,f(x;w)表示RNN网络,G表示最优终止步骤。平衡超参数δ = 0.01可以将学习到的步骤的值范围近似归一化为[0,0.75],从而简化了训练过程。RNN模型使用从训练量中得到的推理结果进行训练。
使用小批量随机梯度下降(SGD)[42]优化器训练RNN变体(原始RNN和LSTM (Long Short Term Memory)[41]),
batch size =100, learning rate = 1e-4, moment is 0.5, epoch=100,花费约为45分钟。
隐藏单元数为64个,RNN层数为2个。
3D U-net(检测胎儿脑landmarks)
Adam优化器,batch size = 1, learning rate = 0.001, moment is 0.5, epoch = 40
受GPU内存限制,超声体积调整为0.4倍用于训练。
特征点的高斯地图作为标准切面生成。
用L2-范数回归损失训练了一个定制的3D U-net [38](检测了胎儿大脑的三个标志,即胼胝体膝、胼胝体压部和小脑蚓部,如图(a)三个红点),所示表示为:

(公式5)
其中N = 3为landmark数,Hi、ˆHi分别表示第i个预测的landmark热图和标准landmark热图。
基于验证集选择了超参数,并在保留的测试集上使用几个指标来评估我们的方法的性能。
对每个具有不同大小的超参数进行了模型训练,并评估了在验证数据集上的性能。
选择了具有最佳验证性能的超参数值作为训练阶段的默认设置。
在本研究中,搜索了三个高影响的超参数,包括Replay Buffer的大小、γ和 。
。
评定标准
使用了三个标准来评估平面定位
空间相似性:
1.两个平面之间的二面角(Ang)

np,ng表示预测平面和目标平面的法线
2.两个平面到原点的欧氏距离差(Dis)

dp,dg表示与体积的距离原点到预测平面,原点到真实平面
(Ang和Dis是基于平面采样函数,即cos(α)x + cos(β)y + cos(γ)z = d,
有效体素大小为0.5 mm3/voxel)
内容相似性:
3.峰值结构相似度(SSIM0)【43】。
并将迭代t中的ADI定义为距离起始平面的距离和角度的累积变化之和,如下

结果
表一是我们提出的方法和其他现有方法在胎儿我们中的比较结果(平均±std,BEST结果以粗体突出显示)。
Ours:RL(WSADT)

表二:我们提出的方法与其他现有方法在我们子宫中的比较结果(平均±std,BEST结果以粗体突出显示)。

表III对胎儿热起始分析的消融研究的比较结果(平均±标准疾病,BEST结果以粗体突出显示)。
 表iv我们子宫热启动分析消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。
表iv我们子宫热启动分析消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。

表v对我们胎儿的终止策略进行分析的消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。
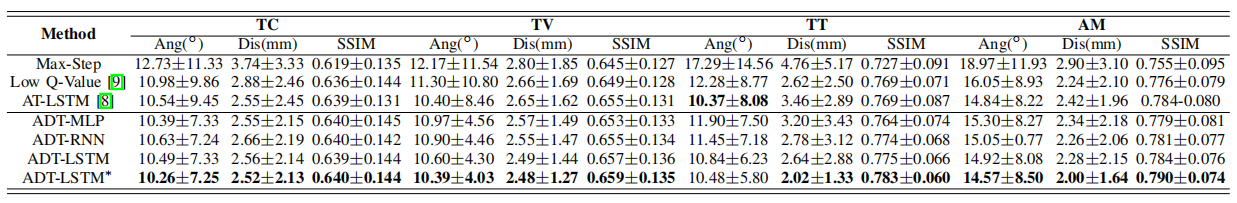
表vi为我们分析子宫终止策略的消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。
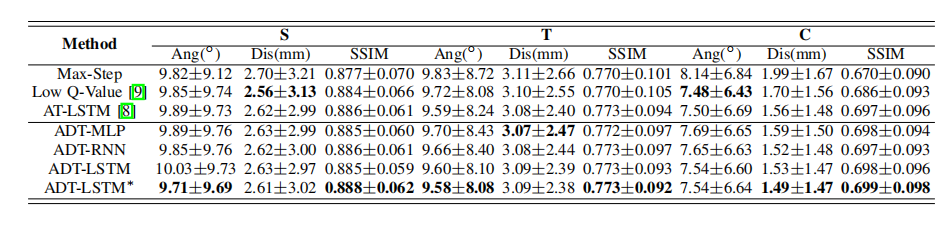 表七、自适应动态终止和主动终止的平均终止步骤
表七、自适应动态终止和主动终止的平均终止步骤
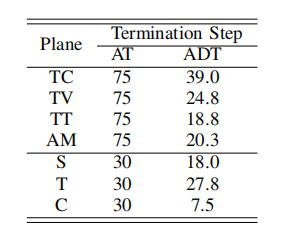
表八:胎儿大脑数据集中LSTM的层数和隐藏量的消融研究。
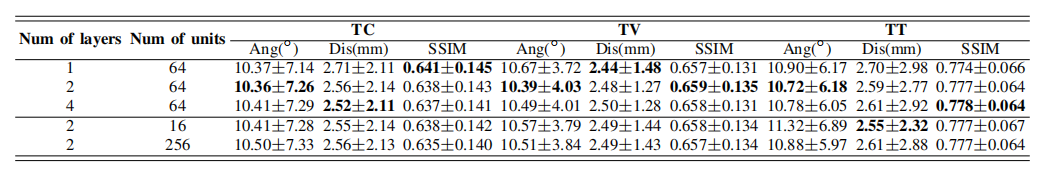
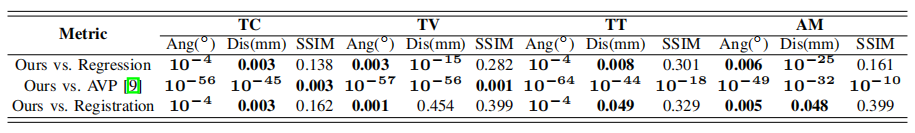
表九胎儿数据集三个性能指标的每个方法和我们的方法之间的两两t检验的p值。粗体的结果表示显著差异
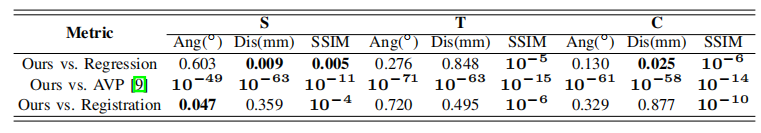
表X子宫数据集中三个性能指标的每个方法和我们的方法之间的两两t检验的p值。粗体结果表示显著差异
表xi的分割性能及临床评估的定量分析

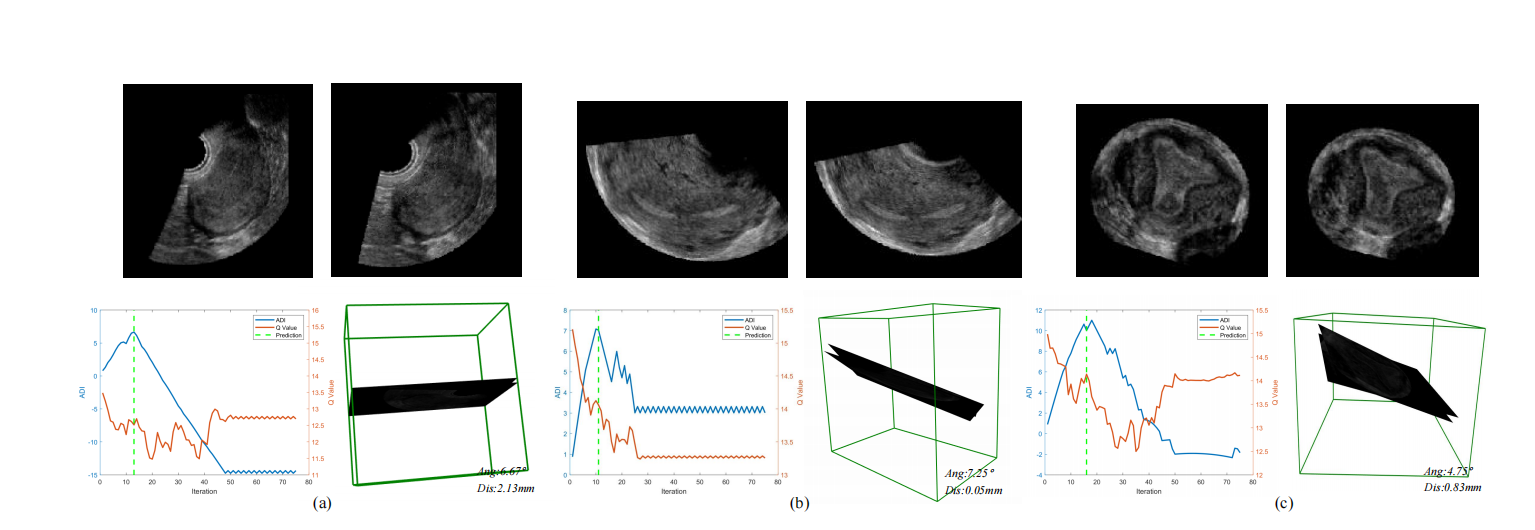
图7。我们的方法在超声胎儿数据集的抽样SPs中的可视化。(a)是经小脑SP,(b)是经心室SP,(c)是经丘脑SP,(d)是腹部SP。对于每种情况,左上角是预测的标准平面,右上角是真实值,左下角是终止模块的推断曲线,右下角是预测平面和真实值的三维空间位置值。

图8。我们的方法在超声子宫数据集的采样SPs中的可视化。(a)为中矢状面SP,(b)为横向SP,(c)为冠状面SP。对于每种情况,左上角为预测的标准平面,右上角为真实值,左下角为终止模块的推断曲线,右下角为预测平面和真实值的三维空间位置。
与最先进的方法进行比较
为了检验我们提出的方法在标准平面定位中的有效性,我们与经典的基于学习的回归方法进行了比较实验,记为回归,目前最先进的自动视图规划方法[9],记为AVP,和我们之前的方法[8],记为RL-US。为了实现公平的比较,我们使用了回归和AVP的默认平面初始化策略,并使用公共实现对所有两个比较模型进行了重新训练。我们还调整了训练参数,以获得最佳的定位结果。如表I和表II所示,可以观察到,我们的方法在几乎所有的指标上都达到了最高的精度。这表明我们的方法在标准平面定位任务中具有优越的能力。
地标标齐模块的影响
为了验证所提方法的地标感知对齐模块的影响,我们比较了该框架在有和没有该框架时的性能。在预回归方法中,我们设置了具有随机启动的代理。平面函数像[9]一样,并选择最低的q值[9]作为终止步骤。Regist方法表示配备了对齐模块,但没有代理搜索的框架。后回归方法表示使用对齐模块进行预热初始化的代理的搜索结果。我们还选择了最低q值终止策略来实现后回归策略,以进行公平的比较。如表III和表II所示,预测方法的准确性明显低于预测方法和后预测方法。这证明了地标感知对准模块能够一致地提高平面检测精度。图5提供了胎儿大脑标志对齐前后的三维空间分布。可以观察到,所有的地标都被映射到一个相似的空间位置,这表明所有的胎儿姿势都是大致对齐的
自适应动态终止的分析
为了证明所提出的自适应动态终止(ADT)策略的影响,我们与现有的流行策略进行了比较实验,如使用最大迭代的终止(Max-Step)、最低Q值(低Q值[9])和使用LSTM的主动终止[8](AT-LSTM)。我们还将我们提出的具有不同主干网络的ADT进行了比较,包括多层感知器(ADT-MLP)、香草RNN(ADT-RNN)和LSTM(ADT-LSTM)。上标∗表示模型用归一化的MSE损失函数(LMSE,等式4)如表V和表VI所示,采用自适应动态终止策略后,agent能够避免陷入较差的局部最小值,并获得了更好的性能。此外,从表七中,我们可以观察到,我们提出的动态终止最多可以节省约67%的推理时间,从而提高了强化框架的效率。
显著差异分析
为了调查不同方法之间的差异是否有统计学意义,我们对我们的方法的结果与回归、AVP [9]、配准进行了配对t检验。对所有的性能指标都进行了这些测试。包括角度,距离和SSIM。我们设置显著性水平为0.05。结果如表IX和x所示。表I-IV和IX-X中的比较和检验结果表明,我们的方法在现有方法(回归、AVP [9])和配准中表现最好。虽然我们的方法优于AT-LSTM [8],但没有显著差异,但我们的方法最多可以节省67 %的推理时间,如表7所示。
来自SP的临床生物特征评估
在本节中,我们将进一步探讨检测到的平面是否能够提供与人工获得的平面获得的一致的准确生物识别,这是更多临床关注的问题。为了获得预测平面(TT和AM)上的数据,我们使用预先训练好的DeepLabv3+ [44]对胎儿头部和腹部进行分割。然后生成胎儿头部或腹周的两个最小椭圆和目标平面上标注的真实值。我们使用三个指标来评估生物测量的性能,包括骰子得分(骰子)、绝对误差(AErrorr)和相对误差(R-Errorr)。如表十一所示,所提方法在Dice评分上取得了良好的性能。同时,本方法的胎儿头围和腹围的绝对误差和相对误差分别为1.125mm、2.05%和3.608mm、3.25%。表XI中的p值也表明,我们预测的生物特征与注释没有显著差异。这显示了与人类水平的性能[45],[46]相似的性能,并表明所提出的方法有潜力应用于实际的临床环境。
定性评价
图7和图8提供了所提方法的可视化结果。它显示了四种随机选择的案例的预测平面、真实值、终止曲线和三维空间可视化。可以观察到,预测在空间上接近,在视觉上与真相相似。此外,该方法可以一致地达到一个理想的停止点。最大迭代终止策略和最低Q值终止策略都未能发现最优终止步骤。
虽然RL在MRI [9]中能有效地定位视野平面,但在3D US中未能定位SPs的定位。没有对齐模块和早期停止设置,AVP需要在巨大的搜索空间中进行代理训练和推理。因此,基于学习的定位方法更容易在有限的搜索空间内定位SP。这可能解释了表三和表四中[9]的性能相对较低的原因。所提出的地标感知对齐模块是基于确切的关注而设计的。它使用刚性配准将所有的体积对齐到相同的图谱空间,这可以像在MRI图像中那样约束环境。此外,我们提出的对齐方法可以看作是在测试US卷时基于预先的代理初始化,它将搜索空间减少到一个细粒度的子空间。
在深度RL中,适当的终止策略是必不可少的,而在迭代搜索过程中,代理经常被困在局部最小值中,因此很难估计最优终止步骤。先前的研究为[7],[9].提出了几种不同的终止策略。然而,如表V和表VI、图7和图8所示,上述实验或先前基于知识的终止策略未能在这一具有挑战性的任务中估计最优终止步骤。同时,之前的研究[9],[8]默认代理在固定的最大步骤终止,导致定位系统的效率低下。我们之前的研究设计了一个基于学习的主动终止,使用RNN来学习q值序列和最优步骤之间的映射。但是,它也需要等待代理来完成推理。相比之下,我们的终止模块允许使用RNN进行动态代理搜索的学习q值曲线与最优终止步骤之间的隐式关系。所得到的RL框架可以实现更准确的、更有效的预测。请注意,这种基于学习的终止策略是一种通用的方法,可以应用于其他类似的任务。
博文相关链接
识别、提取三维超声中标准平面的总结+论文+代码合集_luemeon的博客-CSDN博客
Searching Collaborative Agents for Multi-plane Localization in 3D Ultrasound多智能体强化学习(MARL)定位超声多个标准切面_luemeon的博客-CSDN博客
Agent with Warm Start and Active Termination for Plane Localization in 3DUltrasound基于强化学习的超声标准切面_luemeon的博客-CSDN博客
Agent with Tangent-based Formulation and Anatomical Perception for Standard Plane Localization in 3D_luemeon的博客-CSDN博客
论文References
R
EFERENCES
[1] L. Salomon, Z. Alfifirevic, C. Bilardo, G. Chalouhi, T. Ghi, K. Kagan,
T. Lau, A. Papageorghiou, N. Raine-Fenning, J. Stirnemann, S. Suresh,
A. Tabor, I. Timor-Tritsch, A. Toi, and G. Yeo, “Erratum: ISUOG
practice guidelines: Performance of fifirst-trimester fetal ultrasound scan,”
Ultrasound in Obstetrics and Gynecology
, vol. 41, no. 2, pp. 102–113,
2013.
[2] L. Salomon, Z. Alfifirevic, V. Berghella, C. Bilardo, E. Hernandez
Andrade, S. Johnsen, K. Kalache, K.-Y. Leung, G. Malinger, H. Munoz,
et al.
, “Practice guidelines for performance of the routine mid-trimester
fetal ultrasound scan,”
Ultrasound in Obstetrics & Gynecology
, vol. 37,
no. 1, pp. 116–126, 2011.
[3] A. I. Namburete, R. V. Stebbing, and J. A. Noble, “Diagnostic plane
extraction from 3d parametric surface of the fetal cranium,” in
MIUA
,
pp. 27–32, 2014.
[4] A. W. Moore, “Effificient memory-based learning for robot control,” 1990.
[5] A. G. Barto, R. S. Sutton, and C. W. Anderson, “Neuronlike adaptive
elements that can solve diffificult learning control problems,”
IEEE
Transactions on Systems, Man, and Cybernetics
, vol. SMC-13, no. 5,
pp. 834–846, 1983.
[6] J. C. Caicedo and S. Lazebnik, “Active object localization with deep
reinforcement learning,” in
2015 IEEE International Conference on
Computer Vision (ICCV)
, pp. 2488–2496, 2015.
[7] F.-C. Ghesu, B. Georgescu, Y. Zheng, S. Grbic, A. K. Maier, J. Horneg
ger, and D. Comaniciu, “Multi-scale deep reinforcement learning for
real-time 3d-landmark detection in ct scans,”
IEEE Transactions on
Pattern Analysis and Machine Intelligence
, vol. 41, pp. 176–189, 2019.
[8] H. Dou, X. Yang, J. Qian, W. Xue, H. Qin, X. Wang, L. Yu, S. Wang,
Y. Xiong, P.-A. Heng,
et al.
, “Agent with warm start and active
termination for plane localization in 3d ultrasound,” in
International
Conference on Medical Image Computing and Computer-Assisted Inter
vention
, pp. 290–298, Springer, 2019.
[9] A. Alansary, L. Le Folgoc, G. Vaillant, O. Oktay, Y. Li, W. Bai,
J. Passerat-Palmbach, R. Guerrero, K. Kamnitsas, B. Hou,
et al.
,
“Automatic view planning with multi-scale deep reinforcement learning
agents,” in
International Conference on Medical Image Computing and
Computer-Assisted Intervention
, pp. 277–285, Springer, 2018.
[10] D. Ni, X. Yang, X. Chen, C.-T. Chin, S. Chen, P. A. Heng, S. Li,
J. Qin, and T. Wang, “Standard plane localization in ultrasound by
radial component model and selective search,”
Ultrasound in Medicine
& Biology
, vol. 40, no. 11, pp. 2728–2742, 2014.
[11] X. Yang, D. Ni, J. Qin, S. Li, T. Wang, S. Chen, and P. A. Heng,
“Standard plane localization in ultrasound by radial component,” in
2014 IEEE 11th International Symposium on Biomedical Imaging (ISBI)
,
pp. 1180–1183, IEEE, 2014.
[12] B. Lei, L. Zhuo, S. Chen, S. Li, D. Ni, and T. Wang, “Automatic
recognition of fetal standard plane in ultrasound image,” in
2014 IEEE
11th International Symposium on Biomedical Imaging (ISBI)
, pp. 85–88,
IEEE, 2014.
[13] L. Zhang, S. Chen, C. T. Chin, T. Wang, and S. Li, “Intelligent scanning:
Automated standard plane selection and biometric measurement of early
gestational sac in routine ultrasound examination,”
Medical Physics
,
vol. 39, no. 8, pp. 5015–5027, 2012.
[14] H. Chen, D. Ni, J. Qin, S. Li, X. Yang, T. Wang, and P. A. Heng, “Stan
dard plane localization in fetal ultrasound via domain transferred deep
neural networks,”
IEEE Journal of Biomedical and Health Informatics
,
vol. 19, no. 5, pp. 1627–1636, 2015.
[15] H. Chen, D. Ni, X. Yang, S. Li, and P. A. Heng, “Fetal abdominal stan
dard plane localization through representation learning with knowledge
transfer,” in
International Workshop on Machine Learning in Medical
Imaging
, pp. 125–132, Springer, 2014.
[16] H. Chen, L. Wu, Q. Dou, J. Qin, S. Li, J.-Z. Cheng, D. Ni, and P.-A.
Heng, “Ultrasound standard plane detection using a composite neural
network framework,”
IEEE Transactions on Cybernetics
, vol. 47, no. 6,
pp. 1576–1586, 2017.
[17] W. Huang, C. P. Bridge, J. A. Noble, and A. Zisserman, “Temporal
heartnet: towards human-level automatic analysis of fetal cardiac screen
ing video,” in
International Conference on Medical Image Computing
and Computer-Assisted Intervention
, vol. 10434, pp. 341–349, Springer,
2017.
[18] Y. Gao and J. A. Noble, “Detection and characterization of the fetal
heartbeat in free-hand ultrasound sweeps with weakly-supervised two
streams convolutional networks,” in
International Conference on Medi
cal Image Computing and Computer-Assisted Intervention
, vol. 10434,
pp. 305–313, Springer, 2017.
[19] C. F. Baumgartner, K. Kamnitsas, J. Matthew, T. P. Fletcher, S. Smith,
L. M. Koch, B. Kainz, and D. Rueckert, “Sononet: real-time detection
and localisation of fetal standard scan planes in freehand ultrasound,”
IEEE Transactions on Medical Imaging
, vol. 36, no. 11, pp. 2204–2215,
2017.
[20] J. Schlemper, O. Oktay, M. Schaap, M. Heinrich, B. Kainz, B. Glocker,
and D. Rueckert, “Attention gated networks: Learning to leverage salient
regions in medical images,”
Medical Image Analysis
, vol. 53, pp. 197–
207, 2019.
[21] L. Wu, J.-Z. Cheng, S. Li, B. Lei, T. Wang, and D. Ni, “FUIQA: Fetal
ultrasound image quality assessment with deep convolutional networks,”
IEEE Transactions on Cybernetics
, vol. 47, no. 5, pp. 1336–1349, 2017.
[22] H. Luo, H. Liu, K. Li, and B. Zhang, “Automatic quality assessment
for 2d fetal sonographic standard plane based on multi-task learning,”
arXiv preprint arXiv:1912.05260
, 2019.
[23] Z. Lin, S. Li, D. Ni, Y. Liao, H. Wen, J. Du, S. Chen, T. Wang,
and B. Lei, “Multi-task learning for quality assessment of fetal head
ultrasound images,”
Medical Image Analysis
, vol. 58, p. 101548, 2019.
[24] P. Zhu and Z. Li, “Guideline-based machine learning for standard plane
extraction in 3d cardiac ultrasound,” in
Medical Computer Vision and
Bayesian and Graphical Models for Biomedical Imaging
, pp. 137–147,
Springer, 2016.
[25] S. Nie, J. Yu, P. Chen, Y. Wang, and J. Q. Zhang, “Automatic detection
of standard sagittal plane in the fifirst trimester of pregnancy using 3-
d ultrasound data,”
Ultrasound in Medicine & Biology
, vol. 43, no. 1,
pp. 286–300, 2017.
[26] C. Lorenz, T. Brosch, C. Ciofolo-Veit, T. Klinder, T. Lefevre, A. Cav
allaro, I. Salim, A. T. Papageorghiou, C. Raynaud, D. Roundhill,
et al.
, “Automated abdominal plane and circumference estimation in
3d us for fetal screening,” in
Medical Imaging 2018: Image Processing
,
vol. 10574, p. 105740I, International Society for Optics and Photonics,
2018.
[27] K. Chykeyuk, M. Yaqub, and J. A. Noble, “Class-specifific regression
random forest for accurate extraction of standard planes from 3d
echocardiography,” in
International MICCAI Workshop on Medical
Computer Vision
, pp. 53–62, Springer, 2013.
[28] H. Ryou, M. Yaqub, A. Cavallaro, F. Roseman, A. Papageorghiou, and
J. A. Noble, “Automated 3d ultrasound biometry planes extraction for
fifirst trimester fetal assessment,” in
International Workshop on Machine
Learning in Medical Imaging
, pp. 196–204, Springer, 2016.
[29] A. Schmidt-Richberg, N. Schadewaldt, T. Klinder, M. Lenga, R. Trahms,
E. Canfifield, D. Roundhill, and C. Lorenz, “Offset regression networks
for view plane estimation in 3d fetal ultrasound,” in
Medical Imaging
12
2019: Image Processing
, vol. 10949, p. 109493K, International Society
for Optics and Photonics, 2019.
[30] Y. Li, B. Khanal, B. Hou, A. Alansary, J. J. Cerrolaza, M. Sinclair,
J. Matthew, C. Gupta, C. Knight, B. Kainz,
et al.
, “Standard plane
detection in 3d fetal ultrasound using an iterative transformation net
work,” in
International Conference on Medical Image Computing and
Computer-Assisted Intervention
, pp. 392–400, Springer, 2018.
[31] V. Mnih, K. Kavukcuoglu, D. Silver, A. A. Rusu, J. Veness, M. G.
Bellemare, A. Graves, M. Riedmiller, A. K. Fidjeland, G. Ostrovski,
et al.
, “Human-level control through deep reinforcement learning,”
Nature
, vol. 518, no. 7540, pp. 529–533, 2015.
[32] C. J. Watkins and P. Dayan, “Q-learning,”
Machine Learning
, vol. 8,
no. 3-4, pp. 279–292, 1992.
[33] Z. Wang, T. Schaul, M. Hessel, H. Hasselt, M. Lanctot, and N. Freitas,
“Dueling network architectures for deep reinforcement learning,” in
International Conference on Machine Learning
, pp. 1995–2003, 2016.
[34] K. Simonyan and A. Zisserman, “Very deep convolutional networks for
large-scale image recognition,”
arXiv preprint arXiv:1409.1556
, 2014.
[35] S. Ioffe and C. Szegedy, “Batch normalization: Accelerating deep
network training by reducing internal covariate shift,”
arXiv preprint
arXiv:1502.03167
, 2015.
[36] T. Schaul, J. Quan, I. Antonoglou, and D. Silver, “Prioritized experience
replay,”
International Conference on Learning Representations
, 2016.
[37] R. Huang, W. Xie, and J. A. Noble, “VP-Nets: Effificient automatic lo
calization of key brain structures in 3d fetal neurosonography,”
Medical
Image Analysis
, vol. 47, pp. 127–139, 2018.
[38]
¨
O. C¸ ic¸ek, A. Abdulkadir, S. S. Lienkamp, T. Brox, and O. Ronneberger,
“3d U-Net: learning dense volumetric segmentation from sparse anno
tation,” in
International Conference on Medical Image Computing and
Computer-assisted Intervention
, pp. 424–432, Springer, 2016.
[39] A. Paszke, S. Gross, F. Massa, A. Lerer, J. Bradbury, G. Chanan,
T. Killeen, Z. Lin, N. Gimelshein, L. Antiga,
et al.
, “Pytorch: An
imperative style, high-performance deep learning library,” in
Advances
in Neural Information Processing Systems
, pp. 8026–8037, 2019.
[40] D. P. Kingma and J. Ba, “Adam: A method for stochastic optimization,”
arXiv preprint arXiv:1412.6980
, 2014.
[41] S. Hochreiter and J. Schmidhuber, “Long short-term memory,”
Neural
Computation
, vol. 9, no. 8, pp. 1735–1780, 1997.
[42] L. Bottou and O. Bousquet, “The tradeoffs of large scale learning,”
Advances in Neural Information Processing Systems
, vol. 20, pp. 161–
168, 2007.
[43] Z. Wang, A. C. Bovik, H. R. Sheikh, and E. P. Simoncelli, “Image
quality assessment: from error visibility to structural similarity,”
IEEE
Transactions on Image Processing
, vol. 13, no. 4, pp. 600–612, 2004.
[44] L.-C. Chen, Y. Zhu, G. Papandreou, F. Schroff, and H. Adam, “Encoder
decoder with atrous separable convolution for semantic image segmen
tation,” in
Proceedings of the European Conference on Computer Vision
(ECCV)
, pp. 801–818, 2018.
[45] L. Wu, Y. Xin, S. Li, T. Wang, P.-A. Heng, and D. Ni, “Cascaded fully
convolutional networks for automatic prenatal ultrasound image seg
mentation,” in
2017 IEEE 14th International Symposium on Biomedical
Imaging (ISBI 2017)
, pp. 663–666, IEEE, 2017.
[46] T. L. van den Heuvel, D. de Bruijn, C. L. de Korte, and B. van
Ginneken, “Automated measurement of fetal head circumference using
2d ultrasound images,”
PloS one
, vol. 13, no. 8, p. e0200412, 2018.




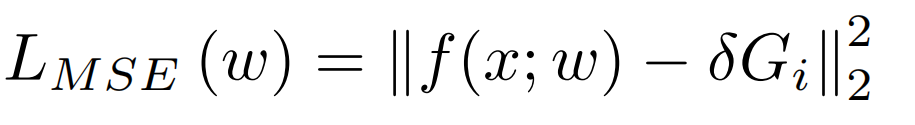
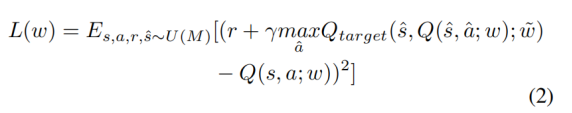
![]() −选择策略[17]的初始首先设置为0.6,
−选择策略[17]的初始首先设置为0.6,


![]() 。
。




 表iv我们子宫热启动分析消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。
表iv我们子宫热启动分析消融研究的比较结果(平均±std,BEST结果以粗体突出显示)。

 表七、自适应动态终止和主动终止的平均终止步骤
表七、自适应动态终止和主动终止的平均终止步骤





