2023年江苏省研究生数学建模科研创新实践大赛A题新型抗癌药物研究模型探索靶向治疗是治疗肿瘤疾病的一种重要方法,它具有针对性强、疗效显著等特点。现有的靶向药物通常针对特定的基因突变靶点,容易出现耐药性。目前,一种由癌症诱发的血管新生作为靶点的靶向药物研究正成为该领域研究的热点。
一、题目重述
原发和转移性肿瘤持续生长的先决条件是肿瘤本身能诱导新的血管生成。定点清除肿瘤新生血管是一种崭新的抗癌策略,该策略通过切断肿瘤赖以生长、转移的营养来源和迁移通道以达到抗癌效果。有证据表明,肿瘤生长、扩散转移与新血管生成密切相关:(a)在肿瘤直径小于2mm时,肿瘤生长缓慢,原发肿瘤仅局部浸润,尚未发生转移,称为“潜伏期”。只有当肿瘤继续生长大于2mm时,微血管逐渐形成,肿瘤实体随之逐渐增大,进而发生扩散和转移;(b)肿瘤实体内微血管数量与肿瘤转移潜能成正相关;(c)某些血管生成素与生长因子,如VEGF、EGF、FGF等通过促进血管生长增加了肿瘤转移的概率;(d)某些血管生成抑制剂能抑制肿瘤细胞生长与转移。基于以上事实,研究血管生成抑制剂以达到阻断肿瘤转移已成为抗肿瘤研究的关键。
目前,依据肿瘤血管发生机制设计的血管抑制剂较多,归纳起来主要有细胞外基质降解抑制剂、粘附分子抑制剂、活化的内皮细胞抑制剂、血管生成因子抑制剂和细胞内信号传导阻断剂等五类。
为了研究某类药物对血管新生的作用,研究人员进行了以下实验:对某种动物使用药物A 诱导其血管新生,加入药物B 作用后发现其具有逆转A 造成的血管新生作用(先加入药物 A,在其作用结束并清洗后,再加入药物B),而药物B 的结构类似物C 对试验动物有明显的血管新生抑制作用。在对四组样品(正常对照组、加药物A 组、加药物B 组和加药物 C)适当处理(包括充分的培养时间和药液清洗)后,进行RNA-seq 测序。本研究希望通过比对正常对照组(没有添加任何药物)、药物A 添加组、药物B 添加组和药物C 添加组的基因表示,研究药物A 诱导血管新生作用、药物B 血管新生逆转作用和药物C 对血管新生的抑制作用机理。
请解决以下问题:1. 针对附件数据,建立基因表达差异的显著性检验模型,并进行相关参数估计。因费用问题实际采集的样本很少,给出提高小样本显著性检验精度的方法;2. 在研究基因表达显著性差异时,一般假设基因表达是独立的。但事实上,生物学功能基因组的表达水平往往具有协同调节特点(inherently coregulated in their expression levels),请建立数学模型刻画基因表达的协同调节作用,并对模型的合理性进行评价;3. 请建立模型,寻找与血管新生直接关联的基因。现有的方法是对表达显著性差异的基因利用 FDR校正以克服检验误差,但这样得到的基因数目通常还有数千个,请结合问题2模型,利用生物学功能基因组协同调节的特点减少敏感基因数目,并针对附件中数据在论文中给出50个最敏感基因。
名称解释:
-
靶向药物:是指被赋予了靶向(Targeting)能力的药物或其制剂。其作用是使药物或其载体能瞄准特定的病变部位,并在目标部位蓄积或释放有效成分。
-
EGF(表皮细胞生长因子):该因子可以在体内促进机体的表皮细胞、上皮细胞等生长分裂代谢,改善细胞生长微环境。
-
FGF(成纤维细胞生长因子):该因子可以调节多种细胞的迁移、增殖、分化、存活、代谢活动和神经功能。
-
VEGF(血管内皮生长因子):该因子可以促进血管形成,增加血管通透性 。
-
血管生成抑制剂:血管生成抑制剂是阻断血管生成的药物,其作用是阻止肿瘤获取养分和氧气。
-
RNA-seq(RNA sequencing):转录组测序技术,常用于检测所有mRNA的表达量差异。该技术利用新一代高通量测序平台对基因组cDNA测序,通过统计相关Reads(用于测序的cDNA小片段)数计算出不同mRNA的表达量,分析转录本的结构和表达水平。
-
FDR(false discovery rate):伪发现率,指错误拒绝的个数占所有被拒绝的原假设个数比例的期望值。
数据说明:
-
样本包括7组实验数据(genes001.xlsx):2个Cont对照组(Cont-1_count_fpkm 和Cont-2_count_fpkm,对未添加任何药物样本测序,并计算基因表达量FPKM);1个添加药物A组(A-1_count_fpkm,直接添加含药物 A 的培养液,经过足够长时间培养后对样本测序,并计算基因表达量FPKM);2个添加药物 B组(B-1_count_fpkm和B-2_count_fpkm,该实验是在添加含药物A培养液,经过适当时间,诱导血管新生后,洗去药液,再加入含药物B的培养液,经过足够长时间培养后对样本测序,并计算基因表达量FPKM);2个添加药物C组(C-1_count_fpkm和C-2_count_fpkm,直接添加含药物C的培养液,经过足够长时间培养后对样本测序,并计算基因表达量FPKM)。
-
Id:基因的ID
-
基因表达量FPKM:Fragments Per Kilobase of transcript per Million mapped reads,其计算公式为
二、问题一的分析
针对附件数据,建立基因表达差异的显著性检验模型,并进行相关参数估计。因费用问题实际采集的样本很少,给出提高小样本显著性检验精度的方法。
对于基因表达差异的显著性检验,可以使用 t t t 检验或者 DESeq2 等差异表达分析工具,下面是一个基于 t t t 检验的简单模型,用于比较两个处理组之间的基因表达差异:
假设我们有两组样本,分别是组 A A A 和组 B B B。对于每个基因,我们有 A A A 组的基因表达值( x 1 x_1 x1, x 2 x_2 x2, …, x n x_n xn)和 B B B 组的基因表达值( y 1 y_1 y1, y 2 y_2 y2, …, y n y_n yn)。我们的零假设( H 0 H_0 H0)是:两组样本的基因表达均值相等,即 μ A = μ B μ_A = μ_B μA=μB。备择假设( H 1 H_1 H1)是:两组样本的基因表达均值不相等,即 μ A ≠ μ B μ_A ≠ μ_B μA=μB。
t检验的统计量可以表示为:
t = ( m e a n ( x ) − m e a n ( y ) ) / s q r t ( ( v a r ( x ) / n ) + ( v a r ( y ) / n ) ) t = (mean(x) - mean(y)) / sqrt((var(x)/n) + (var(y)/n)) t=(mean(x)−mean(y))/sqrt((var(x)/n)+(var(y)/n))
其中,mean(x) 和 mean(y) 分别是组 A 和组 B 的基因表达均值,var(x) 和 var(y) 分别是组 A 和组 B 的基因表达方差,n 是每个组的样本数量。
在小样本情况下,可以考虑使用更稳健的方法来增加显著性检验的精度。例如,可以使用基于重抽样的方法。
二、问题二的分析
数据量差距有点大,先预处理一下吧,归一化。
- 计算基因之间的相关性,可以使用相关系数(如皮尔逊相关系数)或其他距离度量
- 建立基因相关性矩阵
- 基于相关性矩阵,构建网络,节点表示基因,边表示基因之间的相关性
- 可以设定一个相关性阈值来确定是否存在一条边,即相关性大于阈值的基因被连接起来
- 在这个网络中,咱们可以找找看有没有什么搜索算法,来识别出具有高度相关性的基因模块
- 协同调节的基因集合
社区发现算法,论文里提的,图搜索
示例网络:
社区发现的目的也很简单,就是在图中找到一些“潜在的有特定关系的组织”,也就是社区
举一个示例,代码如下:
import matplotlib.pyplot as plt
import networkx as nx
from community import community_louvain
G = nx.karate_club_graph()
com = community_louvain.best_partition(G)
node_size = [G.degree(i)**1*20 for i in G.nodes()]
df_com = pd.DataFrame({
'Group_id':com.values(),
'object_id':com.keys()}
)
df_com.groupby('Group_id').count().sort_values(by='object_id', ascending=False)
colors = ['DeepPink','orange','DarkCyan','#A0CBE2','#3CB371','b','orange','y','c','#838B8B','purple','olive','#A0CBE2','#4EEE94']*500
colors = [colors[i] for i in com.values()]
plt.figure(figsize=(4,3),dpi=500)
nx.draw_networkx(G,
pos = nx.spring_layout(G),
node_color = colors,
edge_color = '#2E8B57',
font_color = 'black',
node_size = node_size,
font_size = 5,
alpha = 0.9,
width = 0.1,
font_weight=0.9
)
plt.axis('off')
plt.show()
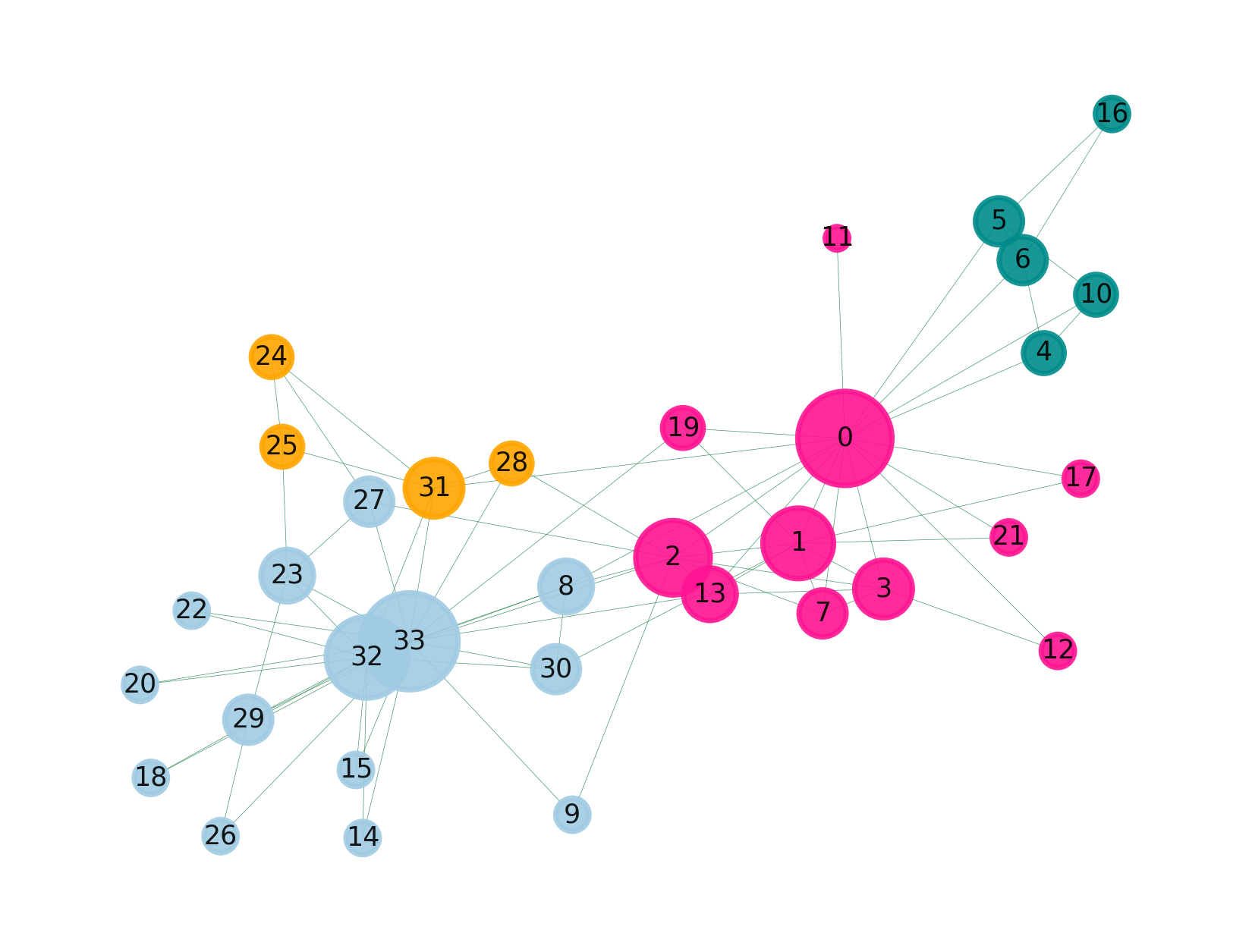
这个数据集也很简单,就是有关联的对应起来:

咱们可以用这个简单的方法来处理,比如设置一个阈值,高于0.3的才称有关联,这样我们就筛选了部分有联系的基因出来,就可以绘制这样的图了。
参考文献:
https://www.jianshu.com/p/b05145d0020a
三、问题三的分析
FDR(False Discovery Rate)校正是一种用于多重比较校正的方法,用于控制在进行多个假设检验时出现的错误发现率。在基因表达分析中,当比较多个基因的表达差异时,FDR校正可以帮助识别那些在显著性检验中具有实际差异的基因,以减少错误发现。
对表达显著性差异的基因进行FDR校正:
第一步:执行显著性差异分析
使用适当的统计方法(例如,t检验、方差分析、Wilcoxon秩和检验等),比较不同条件下的基因表达值,识别出表达显著性差异的基因。这将产生每个基因的p值或其他统计量。
第二步:计算原始FDR值
将所有p值按从小到大的顺序进行排序。然后,计算每个p值对应的FDR值,使用以下公式(公式题目中已给,我就不打了)
第三步:FDR校正
对于设定的期望FDR水平(例如0.05),找到第一个FDR值小于等于该阈值的位置i。然后,所有p值排名在i之前的基因被认为是显著的
第四步:获取显著差异基因
选择根据FDR校正得出的显著差异基因。这些基因被认为在多重比较下仍然具有显著性差异
简单的思路:
- 使用问题2中描述的方法,建立基因表达的协同调节模型,构建共表达网络,识别基因模块。
- 使用已知与血管新生相关的生物学知识,例如文献报道、基因数据库等,来选择与血管新生相关的功能模块。这将帮助缩小关注范围。
- 在功能模块内,对在问题2中鉴定的基因进行进一步筛选,选择与血管新生功能密切相关的基因。这可以基于基因的生物学功能注释、通路分析等。
- 对在步骤4中筛选出的基因进行显著性差异分析,并进行FDR校正,以控制多重比较的错误。
- 基于以上步骤,选择最敏感的前50个基因作为与血管新生直接关联的基因。
补充:
与血管新生相关的基因筛选需要依赖于生物学领域的知识和文献研究。血管新生是一个复杂的生物学过程,涉及多种基因和信号通路的调控。以下是一些可能的筛选规则和方法,用于确定与血管新生相关的基因:
- 回顾已有的文献和数据库,如PubMed、GeneCards、KEGG等,寻找与血管新生过程相关的基因。文献中的研究可以提供关于基因在血管新生中的角色和表达的信息。
- 使用基因功能注释数据库,如Gene Ontology(GO)和Molecular Signatures Database(MSigDB),寻找与血管新生功能关联的基因。这些数据库提供了基因参与的功能、通路和生物过程信息。
- 使用共表达网络分析方法,如WGCNA,构建基因共表达网络,并从网络中识别与血管新生相关的基因模块。(这个好像R语言有)