文本是前文的后续:
《python基于轻量级GhostNet模型开发构建23种常见中草药图像识别系统》
前文主要是在小批量小种类数据集上尝试开发构建基于轻量级CNN模型的中草药图像识别系统,本文的初衷是想要构建一个大类别大数据集的基础,但是无奈发现中草药的种类达到了千余种,且数据采集和人工处理的工作量极大,无奈只好暂时搁置,等待空闲时间再继续投入,这里是爬取构建了40种常见的中草药数据集来开发构建基于MnasNet的识别系统,首先看下实例效果:

MnasNet(Mobile Neural Architecture Search Net)是一种基于神经网络架构搜索(Neural Architecture Search,NAS)的轻量级卷积神经网络模型。通过自动化神经网络架构搜索的方式,找到一种适用于移动设备的高效网络结构。它采用强化学习算法和多目标优化策略来搜索最佳的网络架构。通过对网络的不同部分进行搜索和组合,MnasNet能够在保持较小模型规模的同时,提供较高的性能和准确性。
在设计一个模型搜索算法时,有三个最重要的点:
1、优化目标:决定了搜索出来的网络框架的性能和效率;
2、搜索空间:决定了网络是由哪些基本模块组成的;
3、优化策略:决定了强化学习的收敛速度。
构建原理
MnasNet模型的算法构建原理是通过使用弱连接搜索算法和自动化网络设计方法来构建高效的卷积神经网络模型。MNasNet的搜索空间很大程度上参考了MobileNet v2,通过强化学习的方式得到了超越其它模型搜索算法和其参照的MobileNet v2算法,从中可以看出人工设计和强化学习互相配合应该是一个更好的发展方向。MNasNet中提出的层次化搜索空间可以生成每个网络块都不通的网络结构,这对提升网络的表现也是有很大帮助的,但这点也得益于参考MobileNet v2设计搜索空间后大幅降低的搜索难度。
在弱连接搜索算法中,MnasNet使用了一种基于弱连接的搜索策略,通过在网络的每个位置引入弱连接,然后通过选择性地增加和删除连接来搜索网络结构。这种方法可以显著减少搜索空间并提高搜索效率。
在自动化网络设计方法中,MnasNet使用了一种自动化的网络设计方法来优化网络结构。该方法通过定义一组可行的操作和权重空间,并使用强化学习算法来搜索最优的网络结构。这种方法可以自动地学习网络结构,并在保持高性能的同时减少模型的大小和计算资源的使用。
优点
高性能:MnasNet通过使用弱连接搜索算法和自动化网络设计方法,能够搜索和设计出高性能的卷积神经网络。这些网络在多个图像分类和目标检测任务上展示了较好的性能。
轻量化:MnasNet在网络设计过程中考虑了模型的大小和计算资源的使用。通过自动化网络设计方法,MnasNet能够减少模型的大小和计算量,从而在保持高性能的同时实现轻量化。
可扩展性:MnasNet的自动化网络设计方法具有较强的可扩展性。它可以根据不同的任务和数据集进行自动化的网络设计,并能够适应不同的计算资源限制和应用场景需求。
缺点
训练时间较长:由于MnasNet使用了自动化网络设计方法,需要在大规模的搜索空间中进行搜索和训练,因此训练时间较长。
部署和推理速度较慢:由于MnasNet的网络结构相对较复杂,导致在部署和推理过程中需要更多的计算资源。特别是在计算资源有限的设备上,可能会影响模型的部署和实时推理效果。
MnasNet模型通过弱连接搜索算法和自动化网络设计方法构建了高性能和轻量化的模型。然而,它的训练时间较长,部署和推理速度较慢,需要在实际应用中综合考虑这些因素。
MnasNet核心实现如下所示:
class MNASNet(torch.nn.Module):
def __init__(
self,
alpha: float,
num_classes: int = 1000,
dropout: float = 0.2
) -> None:
super(MNASNet, self).__init__()
assert alpha > 0.0
self.alpha = alpha
self.num_classes = num_classes
depths = _get_depths(alpha)
layers = [
nn.Conv2d(3, depths[0], 3, padding=1, stride=2, bias=False),
nn.BatchNorm2d(depths[0], momentum=_BN_MOMENTUM),
nn.ReLU(inplace=True),
nn.Conv2d(depths[0], depths[0], 3, padding=1, stride=1,
groups=depths[0], bias=False),
nn.BatchNorm2d(depths[0], momentum=_BN_MOMENTUM),
nn.ReLU(inplace=True),
nn.Conv2d(depths[0], depths[1], 1, padding=0, stride=1, bias=False),
nn.BatchNorm2d(depths[1], momentum=_BN_MOMENTUM),
_stack(depths[1], depths[2], 3, 2, 3, 3, _BN_MOMENTUM),
_stack(depths[2], depths[3], 5, 2, 3, 3, _BN_MOMENTUM),
_stack(depths[3], depths[4], 5, 2, 6, 3, _BN_MOMENTUM),
_stack(depths[4], depths[5], 3, 1, 6, 2, _BN_MOMENTUM),
_stack(depths[5], depths[6], 5, 2, 6, 4, _BN_MOMENTUM),
_stack(depths[6], depths[7], 3, 1, 6, 1, _BN_MOMENTUM),
nn.Conv2d(depths[7], 1280, 1, padding=0, stride=1, bias=False),
nn.BatchNorm2d(1280, momentum=_BN_MOMENTUM),
nn.ReLU(inplace=True),
]
self.layers = nn.Sequential(*layers)
self.classifier = nn.Sequential(nn.Dropout(p=dropout, inplace=True),
nn.Linear(1280, num_classes))
self._initialize_weights()
def forward(self, x: Tensor, need_fea=False) -> Tensor:
if need_fea:
features, features_fc = self.forward_features(x, need_fea)
x = self.classifier(features_fc)
return features, features_fc, x
else:
x = self.forward_features(x)
x = self.classifier(x)
return x
def forward_features(self, x, need_fea=False):
if need_fea:
input_size = x.size(2)
scale = [4, 8, 16, 32]
features = [None, None, None, None]
for idx, layer in enumerate(self.layers):
x = layer(x)
if input_size // x.size(2) in scale:
features[scale.index(input_size // x.size(2))] = x
return features, x.mean([2, 3])
else:
x = self.layers(x)
x = x.mean([2, 3])
return x
def _initialize_weights(self) -> None:
for m in self.modules():
if isinstance(m, nn.Conv2d):
nn.init.kaiming_normal_(m.weight, mode="fan_out",
nonlinearity="relu")
if m.bias is not None:
nn.init.zeros_(m.bias)
elif isinstance(m, nn.BatchNorm2d):
nn.init.ones_(m.weight)
nn.init.zeros_(m.bias)
elif isinstance(m, nn.Linear):
nn.init.kaiming_uniform_(m.weight, mode="fan_out",
nonlinearity="sigmoid")
nn.init.zeros_(m.bias)
def cam_layer(self):
return self.layers[-1]
def _load_from_state_dict(self, state_dict: Dict, prefix: str, local_metadata: Dict, strict: bool,
missing_keys: List[str], unexpected_keys: List[str], error_msgs: List[str]) -> None:
version = local_metadata.get("version", None)
assert version in [1, 2]
if version == 1 and not self.alpha == 1.0:
depths = _get_depths(self.alpha)
v1_stem = [
nn.Conv2d(3, 32, 3, padding=1, stride=2, bias=False),
nn.BatchNorm2d(32, momentum=_BN_MOMENTUM),
nn.ReLU(inplace=True),
nn.Conv2d(32, 32, 3, padding=1, stride=1, groups=32,
bias=False),
nn.BatchNorm2d(32, momentum=_BN_MOMENTUM),
nn.ReLU(inplace=True),
nn.Conv2d(32, 16, 1, padding=0, stride=1, bias=False),
nn.BatchNorm2d(16, momentum=_BN_MOMENTUM),
_stack(16, depths[2], 3, 2, 3, 3, _BN_MOMENTUM),
]
for idx, layer in enumerate(v1_stem):
self.layers[idx] = layer
super(MNASNet, self)._load_from_state_dict(
state_dict, prefix, local_metadata, strict, missing_keys,
unexpected_keys, error_msgs)本文中提出的数据集源自自主构建处理,共包含以下类目,如下所示:
三七
人参
佛手片
元胡
厚朴
天南星
天麻
安息香
川芎
巴戟天
当归
木香
朱砂
杜仲
枸杞
桔梗
熊胆
牛黄
玉果
瓜蒌
甘草
生地
白前
白术
白芍
羚羊角
肉苁蓉
苏合香
苦参
茯苓
荜拨
菊花
蔓荆子
贝母
连召
银花
香附
麦冬
黄芪
黄连后续会再此基础上进行扩充类目。
数据实例如下所示:


数据分布可视化如下所示:

整体训练过程loss如下所示:

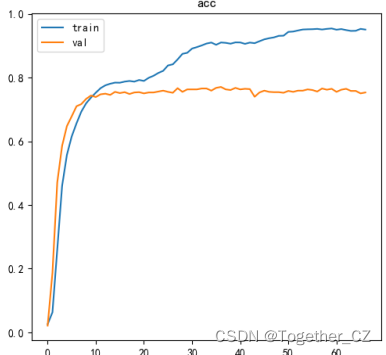
准确率曲线如下所示:
混淆矩阵如下所示:
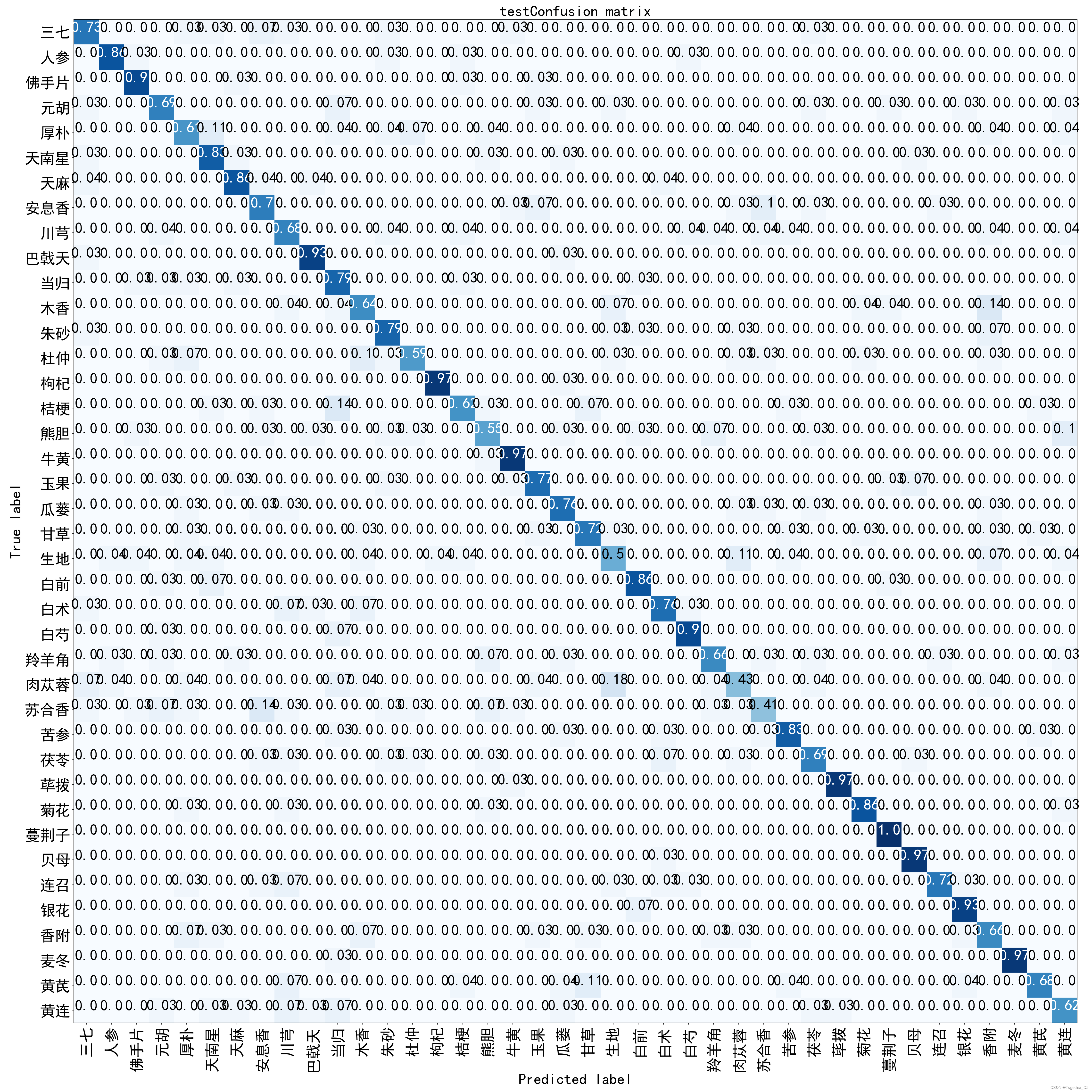
我们对每个类别进行了单独的指标评测,详情如下所示:
+--------+-----------+---------+----------+---------+----------+
| 三七 | 0.70968 | 0.73333 | 0.72131 | 0.71376 | 0.73333 |
| 人参 | 0.89286 | 0.86207 | 0.87719 | 0.87409 | 0.86207 |
| 佛手片 | 0.83871 | 0.89655 | 0.86667 | 0.86312 | 0.89655 |
| 元胡 | 0.66667 | 0.68966 | 0.67797 | 0.66954 | 0.68966 |
| 厚朴 | 0.56667 | 0.60714 | 0.58621 | 0.57557 | 0.60714 |
| 天南星 | 0.70588 | 0.82759 | 0.76190 | 0.75528 | 0.82759 |
| 天麻 | 0.80000 | 0.85714 | 0.82759 | 0.82316 | 0.85714 |
| 安息香 | 0.63636 | 0.70000 | 0.66667 | 0.65735 | 0.70000 |
| 川芎 | 0.57576 | 0.67857 | 0.62295 | 0.61280 | 0.67857 |
| 巴戟天 | 0.87097 | 0.93103 | 0.90000 | 0.89734 | 0.93103 |
| 当归 | 0.58974 | 0.79310 | 0.67647 | 0.66688 | 0.79310 |
| 木香 | 0.64286 | 0.64286 | 0.64286 | 0.63399 | 0.64286 |
| 朱砂 | 0.71875 | 0.79310 | 0.75410 | 0.74745 | 0.79310 |
| 杜仲 | 0.77273 | 0.58621 | 0.66667 | 0.65929 | 0.58621 |
| 枸杞 | 0.96552 | 0.96552 | 0.96552 | 0.96463 | 0.96552 |
| 桔梗 | 0.75000 | 0.62069 | 0.67925 | 0.67179 | 0.62069 |
| 熊胆 | 0.61538 | 0.55172 | 0.58182 | 0.57166 | 0.55172 |
| 牛黄 | 0.84848 | 0.96552 | 0.90323 | 0.90057 | 0.96552 |
| 玉果 | 0.76667 | 0.76667 | 0.76667 | 0.76045 | 0.76667 |
| 瓜蒌 | 0.73333 | 0.75862 | 0.74576 | 0.73911 | 0.75862 |
| 甘草 | 0.77778 | 0.72414 | 0.75000 | 0.74380 | 0.72414 |
| 生地 | 0.53846 | 0.50000 | 0.51852 | 0.50702 | 0.50000 |
| 白前 | 0.83333 | 0.86207 | 0.84746 | 0.84346 | 0.86207 |
| 白术 | 0.78571 | 0.75862 | 0.77193 | 0.76617 | 0.75862 |
| 白芍 | 0.86667 | 0.89655 | 0.88136 | 0.87825 | 0.89655 |
| 羚羊角 | 0.76000 | 0.65517 | 0.70370 | 0.69666 | 0.65517 |
| 肉苁蓉 | 0.52174 | 0.42857 | 0.47059 | 0.45876 | 0.42857 |
| 苏合香 | 0.60000 | 0.41379 | 0.48980 | 0.47913 | 0.41379 |
| 苦参 | 0.82759 | 0.82759 | 0.82759 | 0.82315 | 0.82759 |
| 茯苓 | 0.71429 | 0.68966 | 0.70175 | 0.69422 | 0.68966 |
| 荜拨 | 0.96552 | 0.96552 | 0.96552 | 0.96463 | 0.96552 |
| 菊花 | 0.89286 | 0.86207 | 0.87719 | 0.87409 | 0.86207 |
| 蔓荆子 | 0.87879 | 1.00000 | 0.93548 | 0.93371 | 1.00000 |
| 贝母 | 0.87500 | 0.96552 | 0.91803 | 0.91582 | 0.96552 |
| 连召 | 0.91304 | 0.72414 | 0.80769 | 0.80333 | 0.72414 |
| 银花 | 0.87097 | 0.93103 | 0.90000 | 0.89734 | 0.93103 |
| 香附 | 0.57576 | 0.65517 | 0.61290 | 0.60228 | 0.65517 |
| 麦冬 | 1.00000 | 0.96552 | 0.98246 | 0.98201 | 0.96552 |
| 黄芪 | 0.86364 | 0.67857 | 0.76000 | 0.75477 | 0.67857 |
| 黄连 | 0.66667 | 0.62069 | 0.64286 | 0.63400 | 0.62069 |
+--------+-----------+---------+----------+---------+----------+部分类别精度不高,整体来看还是相对稳定的。