在笔记本上Run起大模型
好久不见的前言
好久没更新了,一是最近一直在研究生物医药大模型相关的内容,二是。相信不止是我,每一位工程师朋友都已经感受到大语言模型带给整个行业的颠覆性改变。最近身边就有不少的小伙伴入坑了大模型技术,而即使仍在观望的同学,也对大模型这一崭新的技术表达了深厚的兴趣。
但说到上手使用大模型,尤其是对于之前没有从事过相关工作的小伙伴来说,门槛是不低的。比如,大模型对基本硬件要求很高,没有大的内存和高性能的GPU就很难玩转,层出不穷的各种新技术和工具更是让人不知道从何入手。
因此,以最近刚刚发布的生物医药大模型——BioMedGPT为例,本教程提供了接触大模型技术的第一步,整理和介绍了在MacBook和Windows笔记本上如何从头开始跑起一个大模型,当然里面用到的技术也同样适用于对其他大模型进行操作。
接下来,我将从如何从零在MacBook和Windows笔记本配置隔离的运行环境,如何从HuggingFace上下载模型、加载模型,以及生成文本(执行推理)几个部分进行介绍。对于有python开发经验的伙伴们,可以选择性阅读。
环境搭建
Mac环境搭建
Mac配置
以本人所使用的MacBook Pro (13-inch, M1, 2020)为例
芯片:Apple M1
内存:16 GB
核总数:8(4性能和4能效)
系统:macOS Monterey,12.6.2
CPU架构:ARM64
conda环境
Anaconda是管理Python环境的强大工具,通过其可以创建、管理多个相互独立、隔离的Python环境,并在环境中安装、管理Python依赖。我们可以使用其免费、最小可用版本MiniConda。
可以在Miniconda ‒ conda documentation找到对应的下载链接和安装方式。

python环境
安装好miniconda以后,我们就可以创建一个Python环境,我们在这里创建了一个名为biomedgpt的python环境,并通过conda activate激活该环境。
conda create -n biomedgpt python=3.10
conda activate biomedgpt
为了运行BioMedGPT-LM-7B,我们需要安装pytorch和transformers。
安装pytorch
Mac上安装Pytoch可以在pytorch官网上找到对应的命令。我们只需要按照下图所示选择对应的版本,然后使用官方给出的安装命令即可。pip install torch torchvision torchaudio

安装transformers
Huggingface提供数以千计针对于各种任务的预训练模型,这些模型被广泛的应用于学术研究当中,transformers是Huggingface开源的一个NLP工具,方便使用者调用这些模型(包括训练、推理、量化等)。

大家可以根据自身的需要,选择适合自己的模型进行训练或微调,也可阅读api文档和源码, 快速开发新模型。我们可以通过pip或者conda直接安装transformers。
pip install transformers
如果pip安装很慢,可以指定清华或者阿里等国内镜像源,下面以清华源为例
pip install transformers -i https://pypi.tuna.tsinghua.edu.cn/simple
Windows环境搭建
conda环境 (可选)
conda提供了两个核心能力:一个是做环境隔离,一个是管理你的安装包。强烈推荐配置conda环境,它可以为你创建一个虚拟环境,最大限度地避免各种环境冲突。
访问 https://docs.conda.io/en/latest/miniconda.html 下载Miniconda:
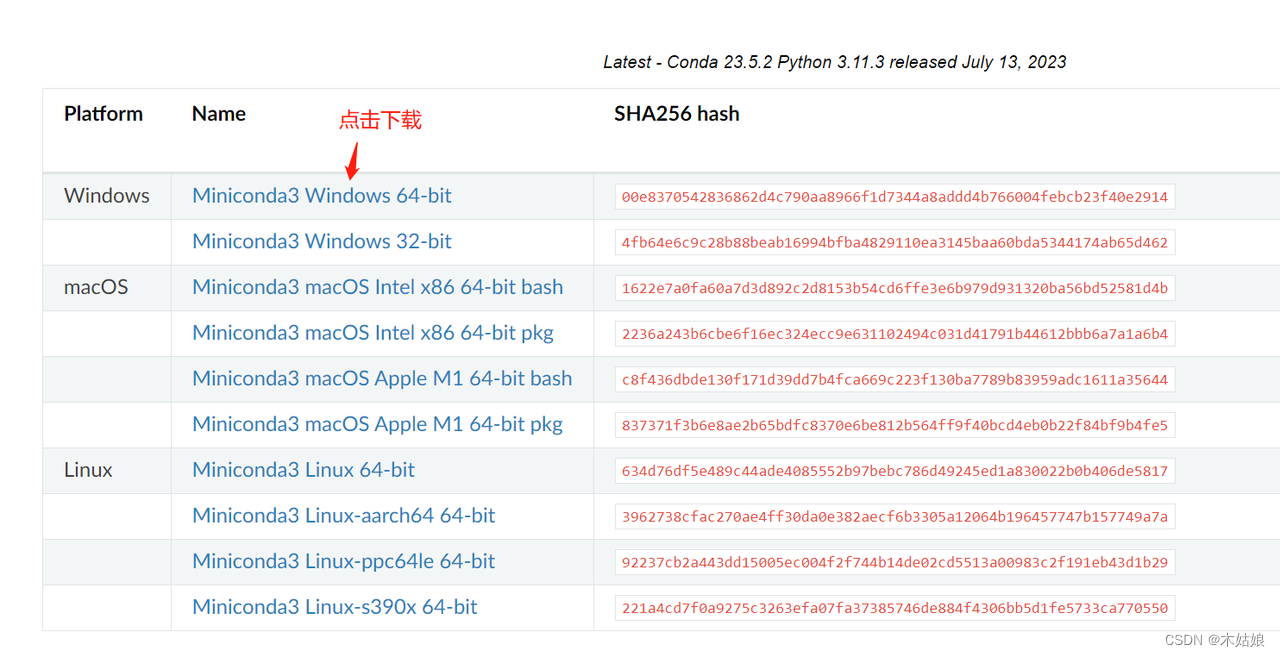
如下图,启动conda命令界面:
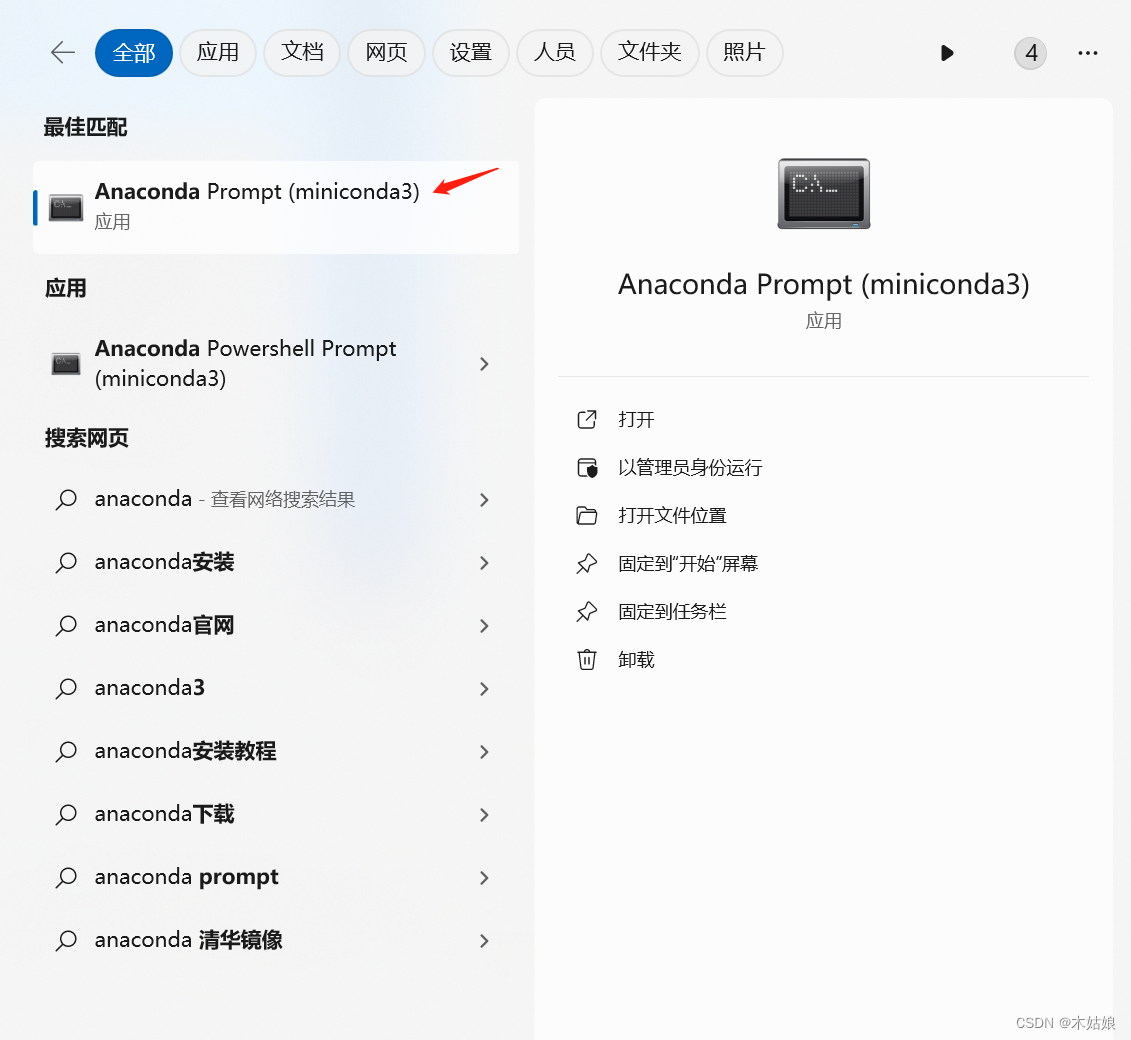
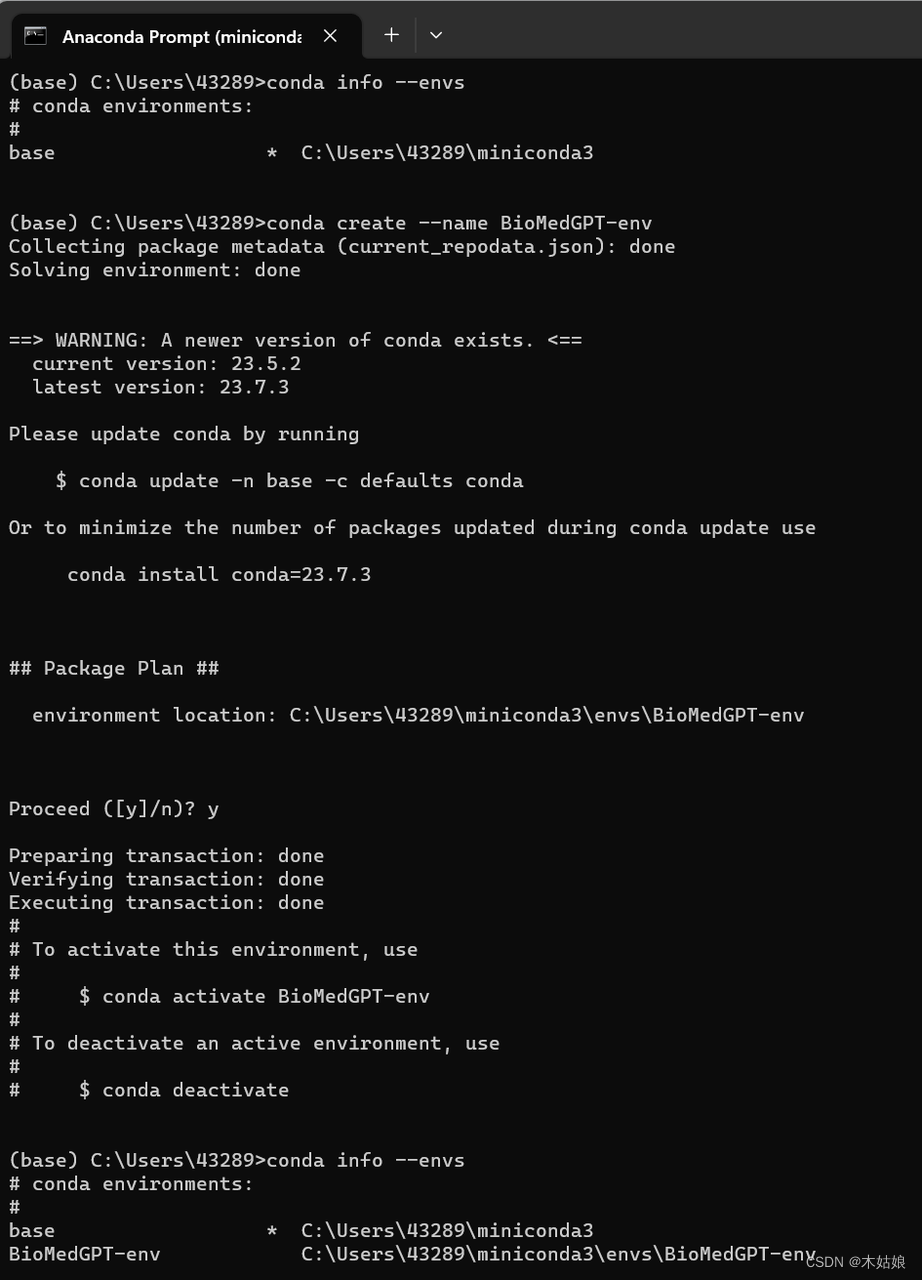
上面截图中的信息表示,初始的时候,conda info --envs命令显示,系统中只有一个名叫“base”的虚拟环境。而在执行了 conda create --name BioMedGPT-env 命令之后,系统中就创建出来一个名叫“BioMedGPT-env”的虚拟环境。下面我们安装所需要的软件包,都会安装到这个虚拟环境下。
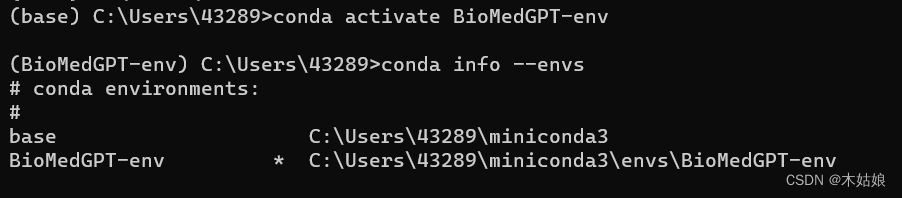
然后使用 conda activate BioMedGPT-env 命令激活这个新的虚拟环境。
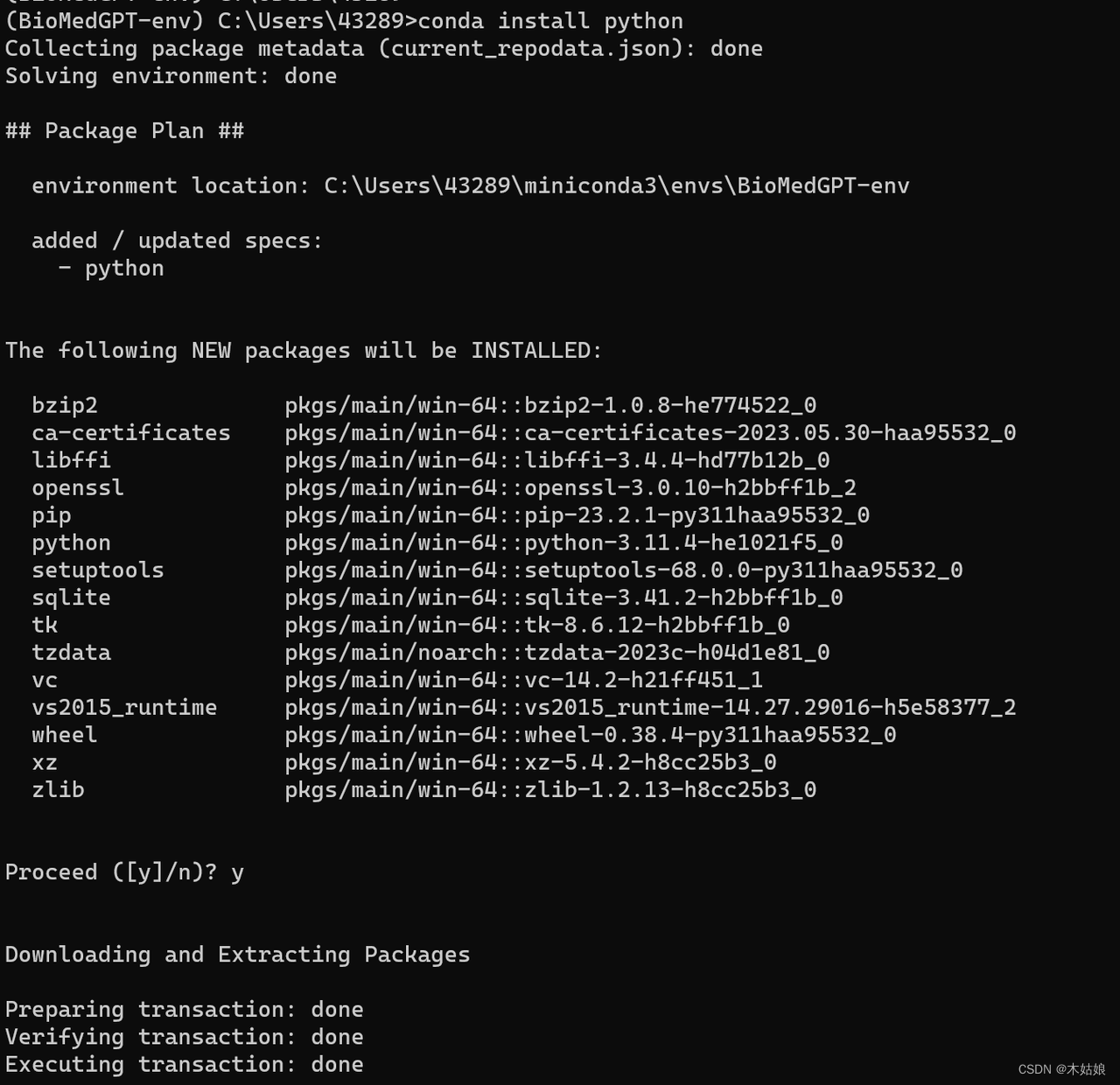
安装python。这一步比较关键,它会确保你的python运行环境从零开始创建,不会受电脑上已经存在的其他python包的影响。
python环境
下面安装一系列需要的python包。本教程为了简单,只需要安装4个python包:torch、transformers、xformers、accelerate。下面执行 pip install 命令进行安装。为了加快从网上下载安装包的速度,用“-i”命令选项指定一下清华的安装镜像地址。
pip install torch transformers xformers accelerate -i https://pypi.tuna.tsinghua.edu.cn/simple
模型下载
BioMedGPT-LM-7B开源在PharMolix/BioMedGPT-LM-7B。

可以通过transformers的AutoModelForCausalLM.from_pretrained自动下载需要的模型和tokenizer。
import transformers
model = AutoModelForCausalLM.from_pretrained("PharMolix/BioMedGPT-LM-7B")
但这种方式速度慢,且经常中断,因此我们也介绍了其他几种下载方式。
方式一:通过git下载
如果你的电脑上还没有安装git,首先安装git。
brew install git
由于模型文件很大,还需要安装Git Large File Storage。
brew install git-lfs
然后可以通过git clone的方式下载。

直接在终端执行git clone https://huggingface.co/PharMolix/BioMedGPT-LM-7B,即可将仓库中所有文件下载到本地。
方式二:直接通过文件链接下载
我们可以进入到PharMolix/BioMedGPT-LM-7B的文件页面。点击文件的下载箭头,就可以直接把文件下载到本地。

需要下载的文件如下,包含了模型和tokenizer所必需的文件。
pytorch_model-00001-of-00003.bin
pytorch_model-00002-of-00003.bin
pytorch_model-00003-of-00003.bin
pytorch_model.bin.index.json
special_tokens_map.json
config.json
generation_config.json
tokenizer.json
tokenizer.model
tokenizer_config.json
方式三:通过huggingface官方提供的模型的下载工具snapshot_download进行下载。
from huggingface_hub import snapshot_download
snapshot_download(repo_id="PharMolix/BioMedGPT-LM-7B")
snapshot_download提供了很多参数,比如你可以通过allow_patterns参数来决定你要下载的文件,resume_download设置为True则可以继续上次中断的下载。更多参数细节请参考官方文档。
from huggingface_hub import snapshot_download
#只下载json文件
snapshot_download(repo_id="PharMolix/BioMedGPT-LM-7B",
allow_patterns="*.json",
resume_download=True)
我们使用Hugging Face提供的transformers框架来加载模型以及必需的其它配置。
模型加载
现在,我们已经准备好了环境和相应的文件,也已经启动了一个jupyter notebook用来编写代码。BioMedGPT-LM-7B是基于meta-llama/Llama-2-7b,在生物医药语料上增量训练得到的,模型的加载方式和llama2-7B模型的加载方式一致。我们可以直接通过transformers进行模型的加载。也可以直接print(model)把模型结构打印出来,具体的模型细节可以查看llama2官网和技术报告。
我们执行以下代码。为了适当暴露加载模型的一些细节,达到教学目的,我们特意单独加载模型和tokenizer,然后采用一个pipeline来把它们串起来(细节见下面的分步骤讲解)。
from transformers import pipeline
from transformers import AutoTokenizer
from transformers import LlamaForCausalLM
import torch
model = LlamaForCausalLM.from_pretrained("PharMolix/BioMedGPT-LM-7B", low_cpu_mem_usage=True)
tokenizer = AutoTokenizer.from_pretrained("PharMolix/BioMedGPT-LM-7B")
pipe = pipeline(task = "text-generation", model = model, tokenizer = tokenizer)

transformers.AutoModelForCausalLM的from_pretrained函数可以从huggingface仓库直接下载模型也可以加载本地下载好的模型。model_path是你存放模型和tokenizer文件的路径。运行上面的代码,我们就成功的在mac笔记本上加载了BioMedGPT-LM-7B模型,我们可以直接打印模型,查看模型的具体信息。如果提示内存不足,建议先关掉其他不需要的进程。
Tokenizer加载
如下图所示(来源于huggingface官方文档),文本在传入到模型到输出结果,需要经过三个步骤。Tokenizer把输入的文本切分成一个一个的token,然后将token转变成向量,Model负责根据输入的变量提取语义信息,输出logits;最后Post Processing根据模型输出的语义信息,执行具体的nlp任务,比如情感分析,文本分类等。
因此,我们需要加载两个核心的对象,一个是模型,一个是tokenizer。在上面代码中,model变量存储了模型本身。而tokenizer的作用是对于模型输入的自然语言文本做预处理(preprocessing)。由于计算机只能对数字进行处理(语言模型也不例外),而我们的输入是自然语言,因此需要一个tokenizer来把文本转成计算机能够处理的数字(最终转成向量的形式,喂给模型)。

tokenizer相关文件和模型文件都在BioMedGPT-LM-7B/文件夹下。其中,tokenizer.json存放了tokenizer的词表,tokenizer_config.json里有tokenizer的相关参数,tokenizer.model则存放着模型参数。 使用tokenizer处理输入的文本得到模型需要的向量表示。

input_ids就是每个token对应的词表id,把其作为模型的输入,得到输出的token id,然后通过tokenizer的decoder方法将output解码成文字。

执行推理
好了,至此,我们终于可以调用模型来生成文本了。我们调用前面的pipe,向模型输入一个问句:“What’s the function of Aspirin?” (翻译:如何设计一款药物?),回答见上图。
结束语
至此,我们成功地在笔记本上加载了7B的大模型,并用来生成了一段文字。注意,由于本教程在笔记本上使用CPU执行,而7B的模型有70亿参数之多,model.generate这步模型推理过程非常耗时,需要耐心等待结果输出(可能需要执行2个小时)。因此,下一篇文章,我们将会和大家一起讨论如何在使用模型的过程中进行加速,包括对模型进行量化和使用GPU。后面我们也会介绍更多的内容给大家,包括如何使用BioMedGPT-LM-7B构建一个医药助手。敬请期待~
下面的二维码,是我们BioMedGPT大模型的官方GitHub地址。本文的教程代码,都在里面的example目录下。后续的教程系列的代码也会持续更新到这个repo中。在手机上阅读的朋友,可以扫码进去star一下,方便后面查阅。
