
期刊:Nature
链接:https://www.nature.com/articles/s41586-022-04862-3
在覆盖了地球表面71%的海洋中,每一滴海水都包含着成千上万种细菌和古菌等微生物,共同构成了全球海洋微生物组(global ocean microbiome)。然而,由于大多数微生物仍未在实验室中实现培养,超过2/3海洋微生物群落的宏基因组无法与已知物种相对应。2022年6月22日,Nature发表了苏黎世联邦理工学院Lucas Paoli等题为“Biosynthetic potential of the global ocean microbiome”的研究论文,通过数十年时间收集并分析了全球215个采样地多个深度的1000多个海洋微生物宏基因组样本,重建了约2.6万个微生物基因组并发现了2700多个未被描述的新物种。结合本研究与已发布的基因组数据,研究人员建立了海洋微生物组学数据库(OMD,Ocean Microbiomics Database),对这一资源的进一步挖掘发现了约4万个生物合成基因簇(BGCs,biosynthetic gene clusters),参与约7000种化合物的生物合成过程。研究还在Ca. Eremiobacterota门中发现了一个BGCs数量丰富且多样性较高的细菌新科,命名为Ca. Eudoremicrobiaceae,并对BGCs中的phospeptin和pythonamide合成途径进行表征后发现了全新的活性物质及酶学机制。本研究突出了微生物组学在深入挖掘天然产物合成与酶学机制中的关键作用,对海洋生态、进化、生物技术与天然产物等领域的研究具有重要意义。
- 研究背景 -
微生物驱动着生物地球化学循环,它们系统发育、代谢与功能的多样性为新类群、酶与生化成分的发掘提供了巨大潜力。在自然环境中,各类天然产物赋予了微生物从信息传递到竞争的多种生理生态功能。同时,天然产物及相关基因编码的合成途径还在生物技术与医疗中具有较高应用价值。对可培养微生物的研究很大程度上助力了相应途径与成分的鉴定,但目前自然界中的大部分微生物仍无法实现培养,极大限制了微生物多样的生物合成途径的应用。为了突破多数微生物无法培养的限制,宏基因组(metagenomics)和单细胞测序技术规避了绝大部分微生物不能培养的缺点,在微生物学研究中得到了广泛应用。利用新一代高通量测序技术(NGS,next-generation sequencing)以特定环境下微生物基因组为研究对象,将测序片段组装成更大的基因组片段并分别重建宏基因组组装基因组(MAGs,metagenome assembled genomes)和单一扩增基因组(SAGs,single amplified genomes),在分析微生物多样性、种群结构、进化关系的基础上,可进一步探究微生物群体功能活性、相互协作关系及与环境之间的关系,发掘潜在的生物学意义。
近年来开展的研究极大扩展了地球上微生物的系统基因组表征,并发现了此前可培养微生物的参考基因组序列(REFs,reference genome sequences)并未包括大多数功能多样的微生物组。将新发现的微生物生理生态功能与相应基因组信息联系起来,对预测具备相关天然产物合成能力的潜在微生物谱系,或确定相关化合物的原始生产者至关重要。例如,已有研究结合了宏基因组与单细胞基因组分析,鉴定了海绵相关的细菌Ca. Entotheonella作为多种候选药物成分的生产者。一直以来,研究人员都致力于在海洋这一地球上最大的生态系统中进行不同微生物群体基因组的解析,但目前仍有超过2/3的海洋微生物宏基因组数据未被解析。因此,海洋微生物组的生物合成及作为新的酶学和天然产物资源库的巨大潜力仍在很大程度上未得到充分探索。
- 研究进展 -
为了在全球范围内研究海洋微生物组的生物合成潜力,本研究首先整合通过培养与非培养方法获得的海洋微生物基因组,建立了一个广泛的系统基因组和基因功能数据库。通过对该数据库的挖掘,发现了不同的生物合成基因簇(BGCs,biosynthetic gene clusters),其中大多数来自于尚未被表征的基因簇家族(GCFs,gene cluster families)。研究进一步鉴定了一个此前未知的基因家族,该家族的BGCs多样性是目前已知的海洋微生物中最高的,研究选取了两种核糖体合成与翻译后修饰多肽(RiPP,ribosomally synthesized and post-translationally modified peptides)途径,功能表征后发现了全新的酶学机制和结构独特的蛋白酶抑制性化合物。
① 全球范围内基因组水平数据资源的建立
首先,本研究致力于建立全球范围内的基因组水平数据资源,在全球215个采样地(纬度范围=141.6°)1-5600 m深度内收集了1038个宏基因组样本,重建了26293个主要为细菌和古菌的宏基因组组装基因组(MAGs,图1),与此前发布的830个MAGs、5969个SAGs和1707个REFs共同组成了34799个基因组。对这一新建立的数据资源进行评估,发现其包括了40-60%的海洋宏基因组数据,与此前仅基于MAGs或SAGs的报道相比,覆盖率增加了2-3倍,在不同深度和纬度范围内的代表性更为一致,因此对海洋微生物组具有更好的代表性。此外,利用基因组分类数据库(GTDB,Genome Taxonomy Database)对所有基因组进行注释,并以95%平均核苷酸同一性进行聚类分析,确定了8304个物种,其中2/3的物种未出现在GTDB,2790个为本研究重建的MAGs中首次发现的。研究还发现不同基因组类型具有高度互补性,分别有55%、26%和11%的物种仅由MAGs、SAGs和REFs组成,MAGs覆盖了水体样本中检测到的全部49个门,而SAGs和REFs分别代表了其中的18个和11个。这一数据库还包括预测的噬菌体和基因组岛等大量未分类的序列片段,研究人员可通过海洋微生物组学数据库(OMD;https://microbiomics.io/ocean/)访问系统分类、基因功能注释以及环境参数信息。

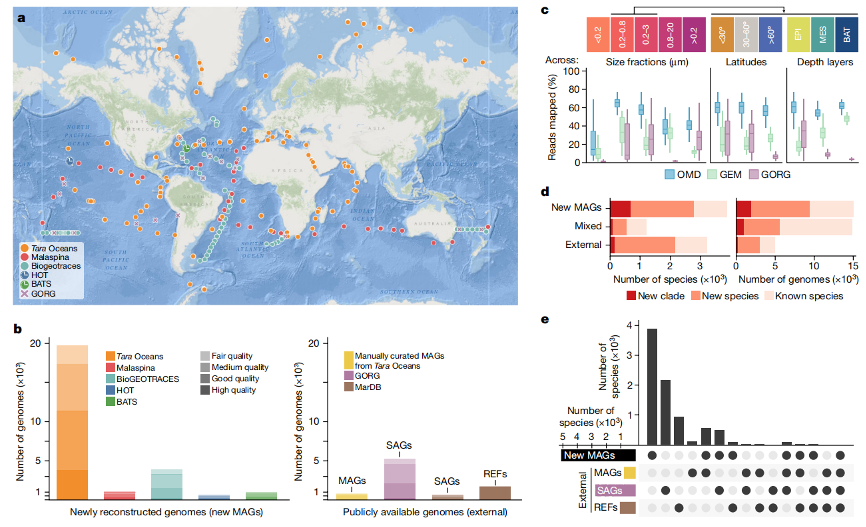
图1 全球范围的MAGs重建填补了海洋系统基因组多样性的空白。
a. 在全球215个采样地采集的1038个海洋宏基因组;b. 利用宏基因组数据重建MAGs,结合此前公布的基因组数据建立海洋微生物组学数据库(OMD);c. 与此前报道的仅基于SAGs或MAGs的数据库相比,OMD将海洋微生物组的基因组比对率提高了2-3倍;d. 物种水平上,OMD中共鉴定出8300个物种(平均核苷酸同一性95%);e. 按基因组类型对物种进行分类,发现MAGs、SAGs和REFs在确定海洋微生物组的系统基因组多样性方面具有高度互补性。
② 生物合成潜力的丰富性和新颖性
随后,为探究海洋微生物组生物合成潜力的丰富性和新颖性,本研究首次在1038个海洋宏基因组中使用antiSMASH对全部MAGs、SAGs和REFs进行检测,预测了39055个BGCs,聚类为6907个非冗余GCFs和151个基因簇家族(GCCs,gene cluster clans)。结果显示,在GCC水平上预测的RiPPs与其他天然产物的多样性较高,其中芳基聚烯、类胡萝卜素、外泌体和铁载体等GCCs在海洋微生物宏基因组中具有广泛的系统基因组分布,表明微生物对海洋环境的广泛适应,包括抗活性氧、氧化和渗透胁迫或铁吸收。这一功能多样性与近期对NCBI RefSeq数据库(BiG-FAM/RefSeq)中约19万个基因组的120万个BGCs分析形成对比,该研究显示非核糖体多肽合成酶(NRPS,non-ribosomal peptide synthetase)和聚酮合成酶(PKS,polyketide synthase)的BGCs占优势。研究还发现,44个(29%)GCCs仅与RefSeq BGCs相关,53个(35%)GCCs仅在MAGs中编码,表明OMD在发掘新化合物方面的潜力。鉴于这些GCCs可能代表高度多样化的生物合成功能,本研究进一步分析了GCFs水平,旨在提供更详细的BGCs聚类以对编码相似的天然产物进行预测。3861个(56%)GCFs与RefSeq数据不重叠,97%的GCFs未在MlBiG中出现,大部分新发现的多样性(3012个GCFs,78%)可对应于预测的萜烯、RiPPs或其他天然产物,其中1815个(47%)GCFs。
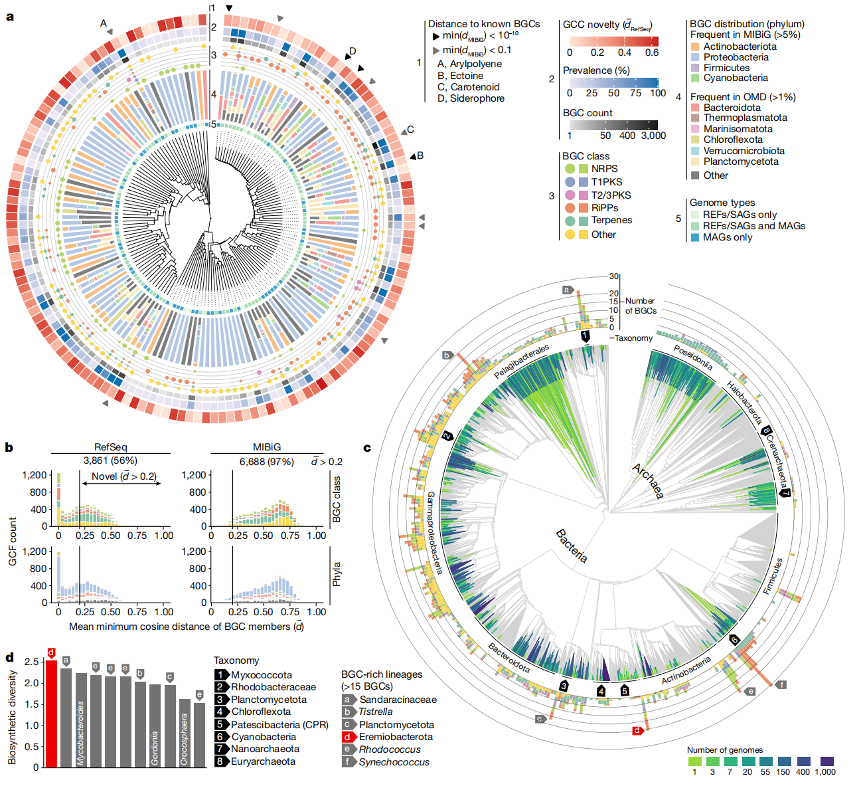
图2 海洋微生物组生物合成潜力的新颖性和系统基因组分布。
a. 基于GCCs的BGC距离的层次聚类;b. GCFs与计算预测(BiG-FAM)和实验验证(MlBiG)的BGCs进行比较,新发现3861个GCFs,其中大多数(78%)编码RiPPs、萜烯和其他潜在天然产物;c. 在1038个海洋微生物宏基因组中检测到的所有OMD基因组均位于GTDB主支上;d. Ca. Eremiobacterota中的未知物种表现出最高的生物合成多样性。
③ 系统发育、生物合成潜力和分布特征
进一步,本研究将海洋微生物基因组对应于标准化的GTDB细菌与古菌系统发育树,并涵盖了它们所编码的潜在生物合成途径。研究发现,重建的MAGs代表了最具生物合成多样性的物种,这些细菌属于未被培养的Ca. Eremiobacterota。进一步分析其系统发育、相对丰度和分布情况表明,Ca. Eudoremicrobiaceae spp.与其他Ca. Eremiobacterota成员相比,具有更大的基因组(8Mb)和更丰富的生物合成潜力(每个物种具有14-22个BGCs),且Ca. Eudoremicrobiaceae spp.在大多数洋盆和表层至4000m水体中数量丰富并普遍存在。此外,Ca. Eudoremicrobiaceae spp.的相对丰度与BGCs表达水平在真核生物富集中最高,这一结果以及部分已知的细胞毒素天然产物生产途径同源的BGCs可能表明与粘球菌(Myxococcus)类似的特殊代谢物生产相关的捕食行为。目前,Ca. Eudoremicrobiaceae为何富集于深层水体或真核生物而非原核生物,仍有待进一步探究。
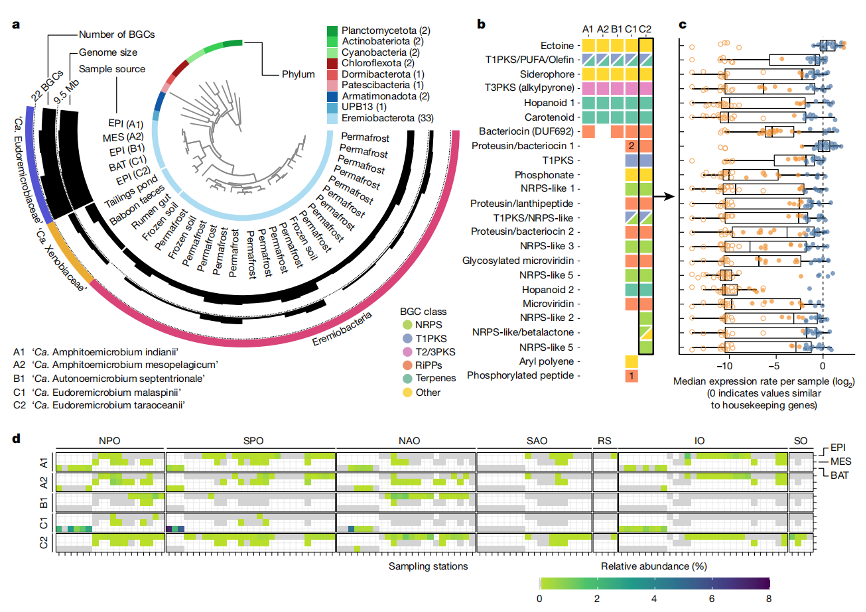
图3 富含BGC家族Ca. Eudoremicrobiaceae的系统发育、生物合成潜力和分布特征。
a. 5个Ca. Eudoremicrobiaceae spp.的系统基因组位置;b. Ca. Eudoremicrobiaceae spp.具有7个核心BGCs;c. 所有Ca. Eudoremicrobium taraoceanii编码的BGCs均在Tara Oceans采集的623个宏转录组中表达;d. 相对丰度显示Ca. Eudoremicrobiaceae spp.在大多数洋盆和表层至4000m水体中数量丰富且普遍存在。
④ 全新的酶和天然产物来源
最后,本研究选取两个Ca. Eudoremicrobium RiPP BGCs进行了分析,验证通过微生物组可以驱动新生物合成途径、酶与天然产物的发现。第一个RiPP途径负责phospeptin的生物合成,与羊毛硫肽类化合物(lanthipeptide)合成酶的脱水结构域同源,但与之不同的是这一途径最终生物合成磷酸化且具有蛋白酶抑制活性的代谢产物。第二个RiPP途径负责proteusin类天然产物pythonamide的生物合成,与常见的proteusin类物质不同的是pythonamide缺乏polytheonamides的NX5N核心基序和landornamides的羊毛硫氨酸环,还首次在RiPP途径中发现了负责N-甲基化的FkbM O-甲基转移酶。
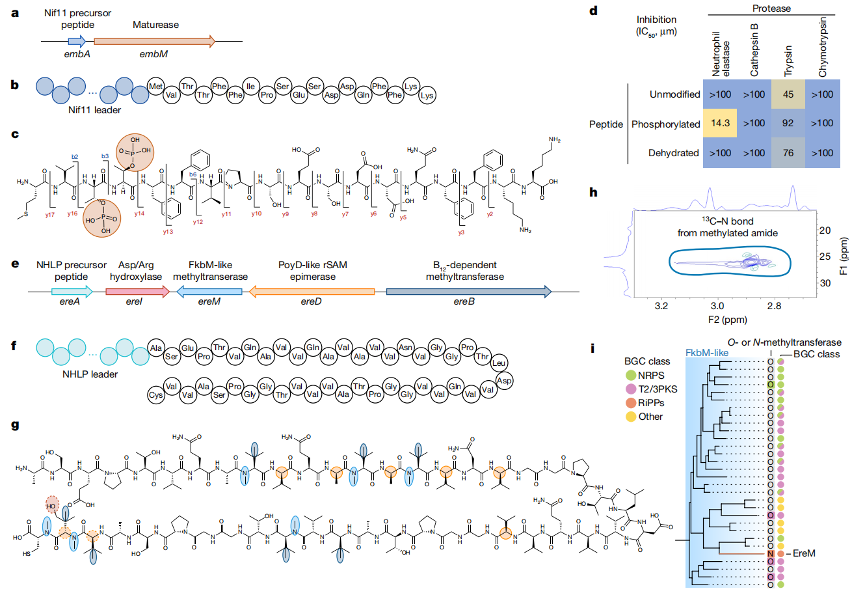
图4 Ca. Eudoremicrobiaceae spp.是一种全新的酶和天然产物来源。
a-c. 针对新型RiPP生物合成簇的体外异源表达与酶学分析;d. 该磷酸化多肽表现出低微摩尔的哺乳动物中性粒细胞弹性蛋白酶抑制;e-g. 另一种新蛋白酶生物合成簇的异源表达揭示了4种成熟酶修饰的一个46氨基酸核心多肽的功能;h. 主链酰胺N-甲基化的核磁共振数据;i. MlBiG 2.0数据库中发现的所有FkbM结构域中FkbM成熟酶的系统发育。
- 研究总结 -
本研究展示了海洋微生物组的生物合成潜力,通过建立海洋微生物组学数据库助力相关研究。研究发现,通过重建MAGs和SAGs可以获得大部分系统基因组和功能新颖性,特别是在尚未被充分探索的微生物群体中,对全新的生物合成途径与酶学机制的挖掘具有重要意义。本研究重点关注了生物合成潜力较高的细菌谱系Ca. Eudoremicrobiaceae,但在其他未知的微生物群体中,可能还存在着许多由BGCs编码的活性物质,将在未来进一步丰富海洋生态进化、生物技术与天然产物等领域的相关研究。
Reference:
Paoli, L., Ruscheweyh, HJ., Forneris, C.C. et al. Biosynthetic potential of the global ocean microbiome. Nature (2022). https://doi.org/10.1038/s41586-022-04862-3
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读