1. 介绍
在定量PCR(也称rt-PCR或qPCR)中,荧光信号是由荧光探针或荧光DNA结合染料(如SYBR Green)产生,其强度与PCR产物分子(扩增子)的数量成正比。每个循环结束时,收集荧光信号强度,通过将荧光强度与循环数作图,qPCR仪生成扩增曲线,其表示在整个PCR过程中产物的累积,通过这个过程,即可实现量化。如果样品中特定的序列(DNA或RNA)丰富,则在较早的循环中观察到扩增,Ct值小;如果序列稀少,则在稍后的循环中观察到扩增,Ct值大。
此SOP将涵盖总RNA提取和两步法逆转录定量PCR(qRT PCR)的操作过程以及数据分析。
2. 注意事项
ü 所有步骤均应在超净工作台上进行,超净台紫外照射至少15min。
ü 所有试剂应为此实验专用。
ü 使用75%乙醇或其他去污剂擦拭工作台,避免表面污染。
ü 进入荧光定量PCR 操作实验室后需要佩戴一次性无菌口罩、无菌乳胶手套、实验中尽量避免与同事交谈,防止PCR 模板污染。
ü 实验中使用各种规格的离心管,枪头及配制试剂和溶解沉淀用的水,均应焦碳酸二乙酯(DEPC)处理后使用,以去除吸附的RNA酶污染。
ü 使用Trizol试剂时,避免接触皮肤或衣服。避免吸入蒸气。
ü qPCR反应需要qPCR级水,试剂和试管。请务必使用正确类型的qPCR光学PCR管和盖子。不可用普通的PCR管。
ü 加样前,先布局,规划加样顺序以避免交叉污染。
ü 所有实验应均应设置无模板对照,其中包含除DNA或cDNA样品以外的所有反应组分。
ü 先配制qPCR反应混合液,减少误差。
ü 加样后上机前,务必通过瞬离将反应液离至管底,并消除溶液中的气泡,因为气泡会干扰荧光检测且降低酶活性,从而降低扩增效率。
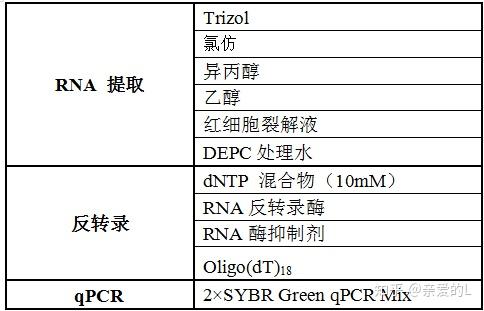
3. 试剂
4. 操作流程
4.1 总RNA提取
A组织样本:迅速将新鲜的组织切成合适的大小,在液氮中充分研磨后加裂解液裂解,或直接加入裂解液后用匀浆器匀浆。每30-50mg组织加1ml Trizol。
B全血样品:加入3-5倍体积的红细胞裂解液到血样中,室温孵育10min,室温3500rpm离心6min。弃上清,原每2-3ml全血样品加1mL Trizol。
C细胞样品:悬浮液中生长的细胞,通过离心收集细胞后,每1×105〜106细胞加入1mL Trizol,移液器吹打混匀,直接裂解或-80℃储存一个月。贴壁生长的细胞,有两种处理方法。一种是移除培养基后,将1mL Trizol直接加入直径3.5cm的培养皿中裂解细胞。或是先用胰蛋白酶将贴壁细胞消化下来并收集后,再加入1mL Trizol进行裂解。(Note:加入Trizol前, PBS充分洗涤细胞去除残余培养基。)
4.1.1 直接法
1)加入1ml Trizol试剂,混匀,冰上孵育10min。
2)4℃,12000rpm离心10min,小心转移上清液到不含颗粒的新EP管中。
3)加入200ul氯仿,剧烈振荡15秒,室温孵育5min。
4)4℃,12000rpm离心15min。混合液分三层,包括最下面的苯酚 - 氯仿有机相,中间相和上层水相。小心转移上层水相到新的EP管(大约60%体积的Trizol),不要吸取中间相,可留下少量上层液体!(Note:质量比数量更重要!)
5)加入等体积的异丙醇,轻轻地颠倒混匀约10次,室温放置10min。
6)4℃,12000rpm离心10min。弃上清,1ml 75%乙醇洗涤RNA沉淀两次。
7)4℃,12000rpm离心5min,弃上清,室温下风干5-10min(不要太干,过度干燥会导致RNA难溶解!)。将RNA溶于15μl-50μlDEPC水中。
8)立即进行反转录反应,或先-80℃长期保存。
4.1.2 离心柱法(依据RNA提取试剂盒说明书)
1)4.1.1中步骤1)—4)。
2)加入1/2体积的无水乙醇,轻柔颠倒混匀。如有透明的纤维状物体,不影响下游步骤。
3)将所有裂解物/乙醇混合物转移至离心柱,置于2mL,静置2min。4℃,12000rpm离心3min,弃废液。
4)加入500uL洗涤液,静置2min。4℃,12000rpm离心30秒,弃废液。重复一次。
5)4℃,10,000rpm离心2min以除去残留的乙醇。
6)将离心柱置于新的RNase-free EP管中,加入30ul-50ulDEPC水,静置5min, 12000rpm离心2min收集。
7)立即进行反转录反应,或先-80℃长期保存。
4.2 反转录
4.2.1 RNA纯度和完整度鉴定
紫外分光光度法测定RNA浓度和纯度
在pH8.0的TE缓冲液(在酸性缓冲液中,Abs 260nm:280nm比值会偏低)稀释RNA,并在260nm和280nm下测量吸光度。
RNA 浓度= 40ug/mL×稀释倍数×Abs 260nm
一般Abs 260nm:280nm比率应该是1.7-2.1,说明RNA的纯度较好。 RNA产量会根据材料的类型和数量以及储存条件的不同而变化。
RNA条带完整度测定
为了评估RNA的完整性,取部分样本做琼脂糖凝胶电泳。完整的RNA条带包括28S,18S和5S核糖体RNA。通常,28S / 18S> 2意味着RNA具有较高的完整度。
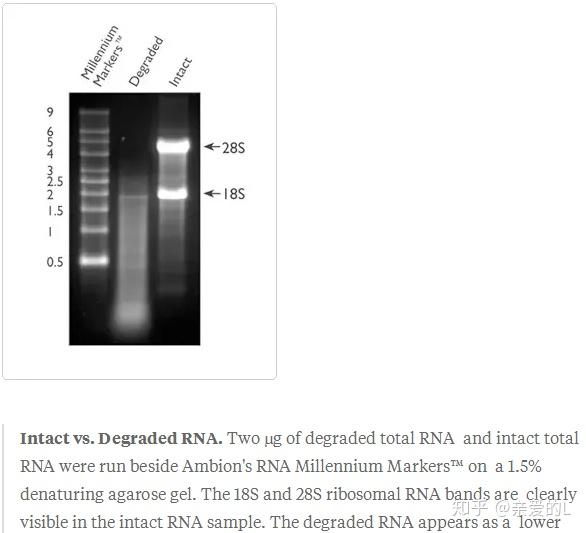
Figure 1
4.2.2 反转录
准备反应主混合物。 每个反应:

PCR条件:42℃孵育40min,85℃加热5min,4℃保存。
将第一链cDNA储存在-20℃。
4.3 引物设计
在Primer5.0软件上设计特异引物。
引物设计标准
1)PCR扩增子长度:50-180bp;
2)引物长度17-25bp;
3)GC含量:45-55%;
4)Tm:58〜68℃,引物对间差异不大于2℃;
5)碱基要随机分布,尽量均匀。3′端要避开AT,GC rich的区域,避开T/C或A/G连续结构(2-3个)。引物自身和引物间不能存在互补。引物3’末端为G或C;
6)避免DNA污染,跨外显子接头区;
7)至少设计2-3对引物,预实验筛选最佳引物;
8)将所有设计的引物在NCBI数据库中比对以确定它们对靶基因特异性。
4.4 Q -PCR 反应

(Note: 一般使用cDNA模板的10倍稀释液加入反应体系,因为过高浓度的反转录体系残留如逆转录酶和相关缓冲液组分会抑制DNA聚合酶活性,从而降低扩增效率。)
扩增循环:
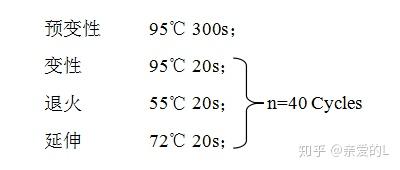
熔解程序:
扩增循环结束后,降温至60℃,然后加热升温至95℃变性DNA产物。
NOTE:
变性 - 高温孵育将dsDNA变成成单链,并松弛ssDNA中的二级结构。 通常DNA聚合酶可承受的最高温度为95℃。如果模板GC含量高,可适当延长变性时间。
退火 - 退火期间,互补序列结合,一般使用低于引物的Tm 5℃作最适退火温度。
延伸 - 70-72℃,DNA聚合酶活性最佳,并且以每秒100个碱基的速率延伸。当qPCR中的产物长度较小时,此步骤可与退火在60℃同步进行。
5. 数据分析
5.1 主要名词
基线 实时PCR反应的基线是指在PCR的初始循环期间的信号水平,通常是3至15的循环之间,此时荧光信号几乎没有变化。此时低信号可以认为是背景或反应的“噪音”。基线应该设置应考虑,在消除早期扩增循环中背景的同时,不覆盖掉明显非背景的扩增信号。当比较不同的qPCR反应或实验时,应定义同一基线。
阈值 qPCR的阈值代表的是明显超出基线的扩增信号水平,用以区分明确的扩增信号和背景。通常,qPCR仪器自带软件会自动将阈值设置为基线荧光值标准偏差的10倍。
Ct Ct是指荧光信号超过阈值时对应的循环数。Ct值与目的基因的起始量成反比,可用于计算DNA初始拷贝数。随着模板量减少,检测到显着扩增时的循环数会增加。
扩增曲线 图2显示的是典型的qPCR扩增曲线。早期循环中,荧光信号几乎没有变化。随着反应的进行,每个循环的荧光水平开始显著上升,被称为指数期。一般在在指数期的早期阶段而不是后期的平台期进行定量分析。在指数阶段开始时,所有的试剂仍然是过量的,DNA聚合酶仍然高效,并且扩增产物量还较低,不会在退火时与引物竞争。这些因素都有助于更准确的定量。
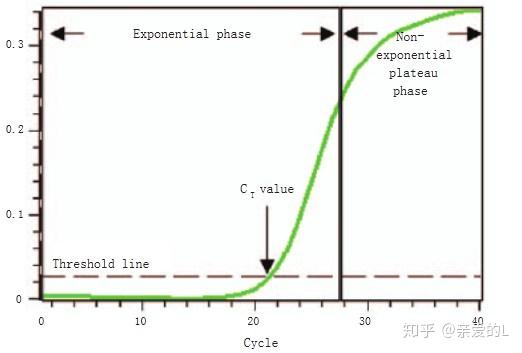
Figure 2
熔解曲线 熔解曲线描绘了随着反应温度升高,掺入染料分子的dsDNA解离成单链DNA(ssDNA)时的荧光变化。例如,当结合SYBR™ Green I染料的dsDNA被加热时,达到解链温度(Tm)后,由于双联DNA链解离而释放出染料,荧光信号会急剧降低。以荧光信号随时间变化作图可得到图3A,然后以ΔF/ΔT(荧光变化/温度变化)值与温度作图,可得到熔融动力学的清晰视图(图3B)。可以根据熔解曲线来判断qPCR反应的特异性,特异性的扩增的溶解曲线会是一个紧密的单峰。
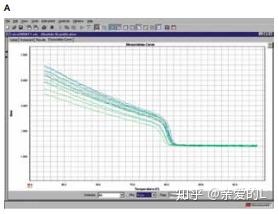

Figure 3
href="">内参 为了实现对目标基因的精确分析,通常使用管家基因(如β-肌动蛋白)来优先计算靶基因的相对表达量,以消除不同组别间因总RNA量的不同而形成的目的基因的表达假性差异。理想情况下,管家基因的表达水平是不随样本类型或是组别处理方法的不同而变化的,应该是常量。
5.2 相对定量
相对定量,不像绝对量化严格,应用于大多数基因表达水研究。相对定量关注的并非某个基因的绝对表达量,而是同一基因在样本间的表达差异(如实验组和对照组)。结果以处理组相对于未处理组的表达差异倍数表示。这种分析方法不需要测定精确的拷贝数,重点是在与未处理样品组相比的表达变化上。
相对定量算法—ΔΔCt ΔΔCt方法是比较组别间同一基因表达差异的常用方法。通过将目的基因Ct(Ct 目的)与自身内参Ct值(Ct 内参)进行比较,可得到每一组别的ΔCt,再将实验组与对照组进行计算得到ΔΔCt。ΔΔCt的大小关联目的基因表达水平的倍数差异大小。
倍数差异 = 2–ΔΔCt
ΔCt 实验组 – ΔCt 对照组 = ΔΔCt
Ct 目的 实– Ct 内参 实 = ΔCt 实验组
Ct 目的 对– Ct 内参 对照 = ΔCt 对照组
6. 疑难
a) RNA 得率低
Ø 样品裂解不完全
适当选择起始材料的量,加入合适体积的裂解液。
Ø 匀浆不充分
减少材料的数量; 根据材料类型选择合适匀浆方法; 组织样本适宜切成小块后,在Trizol试剂中均质,充分裂解。
Ø 用于分光光度分析的稀释液PH值太低
使用TE(PH 8.0)作为稀释液进行读数测定。
b) RNA降解:
Ø RNA酶污染
戴上手套操作,勤换手套; 耗材与试剂均应作RNase-free处理; 操作中减少EP管的开盖次数与时间。
Ø 样品处理不当
新鲜组织收获后,立即冻结在-70°C或液氮中; 也可以在Trizol试剂中均质后,-70℃下储存。
c) Abs260/Abs280 比值低
Ø 酸性pH降低吸光度比率
使用TE缓冲液(pH 8.0)作为稀释液进行测定。
Ø 水相被酚相污染
小心转移上层水相,避免吸入中间相,可留少许上层相不吸取。
d) DNA 污染
Ø Trizol加少了,造成DNA /核蛋白复合物与RNA水相分离不完全
适当选择起始材料的量,加入合适体积的裂解液。
Ø 水相被苯酚相污染
小心转移上层水相,避免吸入中间相,可留少许上层相不吸取。
e) 下游反应中RNA表现不佳
Ø 乙醇或盐类污染
溶解RNA之前,注意洗涤沉淀,并通过空离和风干除去残留。
f) 熔解曲线有杂峰
Ø 引物二聚体
1. 提高退火温度;
2. 降低引物浓度,甚至调整正向引物和反向引物的比例。 一般引物最终浓度设定为200 nM,但如有必要,可降低至60 nM;
3. 镁离子的浓度最好在3mM左右。 高于这个浓度,引物二聚体更易形成;
4. 重新设计引物并评估它们的二聚化倾向。
Ø 非特异性扩增
重新设计引物并通过预实验评估特异性。
Ø cDNA模板讲解
模板避免反复冻融,可适当分装保存,每次取用一小管。
g) 无模板对照有扩增
Ø 引物二聚体
解决方法如上
Ø 加样错误
Ø 核酸污染
1. 使用工作空间的清洁,使用去污剂等擦拭工作台表面;
2. 筛查是否有试剂被核酸污染;
h) Ct值过大
Ø 乙醇或盐类遗留物的抑制
溶解RNA之前,注意洗涤沉淀,并通过空离和风干除去残留。
Ø 扩增效率差
1. 在扩增产物单一情况下,提高镁离子浓度至6mM可以提高扩增效率效率。
2. 根据引物的Tm值设计反应条件(特别是退火温度)是有利的,并且设计引物时应应使正反向引物具有相近的Tm值。
Ø 目标基因低表达
一般基因表达分析需要1-100ng cDNA,但是如果样品中的目的基因丰度过低,则可能需要更多的cDNA样本。 如果不确定目的基因的表达水平,可通过查阅文献或NCBI Unigene数据库得到不同样本中的表达丰度。
i) 无扩增信号
低表达,反转录,或定量仪器问题
文章来自:无锡菩禾生物医药技术有限公司
参考来源: