Несколько дней назад одноклассник спрашивал о реализации метода в статье.После прочтения этой статьи
qPCRв основном чистое письмо за исключением проверки.Сегодня попробую воспроизвести. Повторно появляется рандомно.Если данные чтения не хорошие,можно сдаться.Надеюсь всем понравятся,смотрят и вперед в поддержку.

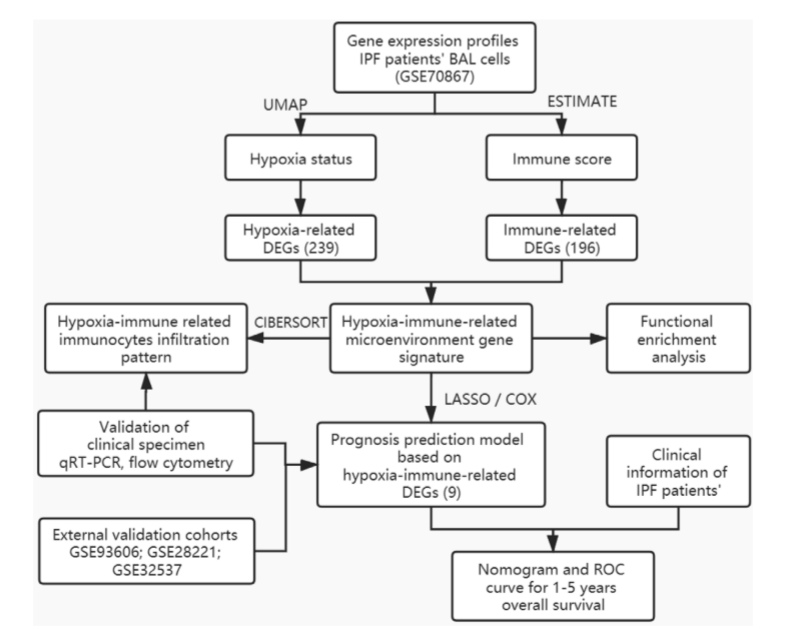
文章标题: Исследование сигнатуры гена микроокружения, связанного с гипоксией, и модель прогнозирования идиопатического легочного фиброза.
дои: 10.3389 / фимму.2021.629854
процесс
Демонстрационные данные и получение кода
Поставьте лайк, прочитайте эту статью, поделитесь ею со своим кругом друзей 集赞10个и 保留30分钟отправьте снимок экрана в WeChat, чтобы mzbj0002получить ее. 2022 VIP-члена получат его бесплатно .
Каноэ Notes 2022 VIP-проект
права и интересы:
Примеры данных и кода всех твитов в Canoe Notes в 2022 году (включая большую часть 2021 года).

Группа обмена научными исследованиями Canoe Notes .
Покупка за полцены
跟着Cell学作图系列合集(бесплатный учебник + коллекция кодов) | Следуйте за Cell, чтобы научиться рисовать коллекцию серий .
ПОТЕРИ:
99р/чел . Вы можете добавить WeChat: mzbj0002перевести деньги или дать вознаграждение прямо в конце статьи.

Загрузка геоданных
GSE70866Есть два GPL, которые необходимо извлечь и аннотировать отдельно.
rm(list = ls())
BiocManager::install("GEOquery")
library(GEOquery)
eSet <- getGEO(GEO = 'GSE70866',
destdir = '.',
getGPL = F)
# 提取表达矩阵exp
exp1 <- exprs(eSet[[1]]) #GPL14550
exp2 <- exprs(eSet[[2]]) #GPL17077
exp1[1:4,1:4]
dim(exp1)
dim(exp2)
#exp = log2(exp+1)
# 提取芯片平台编号
gpl1 <- eSet[[1]]@annotation
gpl2 <- eSet[[2]]@annotation
gpl1
gpl2
## GPL注释
library(devtools)
install_github("jmzeng1314/idmap3")
## 下载后本地安装
## devtools::install_local("idmap3-master.zip")
library(idmap3)
ids_GPL14550=idmap3::get_pipe_IDs('GPL14550')
head(ids_GPL14550)
ids_GPL17077=idmap3::get_pipe_IDs('GPL17077')
head(ids_GPL17077)Матрицы экспрессии генов были аннотированы и объединены.
library(dplyr)
exp1 <- data.frame(exp1)
exp1$probe_id = row.names(exp1)
exp1 <- exp1 %>%
inner_join(ids_GPL14550,by="probe_id") %>% ##合并探针信息
dplyr::select(-probe_id) %>% ##去掉多余信息
dplyr::select(symbol, everything()) %>% #重新排列
mutate(rowMean =rowMeans(.[grep("GSM", names(.))])) %>% #求出平均数
arrange(desc(rowMean)) %>% #把表达量的平均值按从大到小排序
distinct(symbol,.keep_all = T) %>% # 留下第一个symbol
dplyr::select(-rowMean) #去除rowMean这一列
exp2 <- data.frame(exp2)
exp2$probe_id = row.names(exp2)
exp2 <- exp2 %>%
inner_join(ids_GPL17077,by="probe_id") %>%
dplyr::select(-probe_id) %>%
dplyr::select(symbol, everything()) %>%
mutate(rowMean =rowMeans(.[grep("GSM", names(.))]))
arrange(desc(rowMean)) %>%
distinct(symbol,.keep_all = T) %>%
dplyr::select(-rowMean)
exp = exp1 %>%
inner_join(exp2,by="symbol") %>% ##合并探针信息
tibble::column_to_rownames(colnames(.)[1]) # 把第一列变成行名并删除
# 先保存一下
save(exp, eSet, file = "GSE70866.Rdata")
# load('GSE70866.Rdata')
# install.packages("devtools")
# 提取临床信息
pd1 <- pData(eSet[[1]])
pd2 <- pData(eSet[[2]])
## 筛选诊断为IPF的样本
pd1 = subset(pd1,characteristics_ch1.1 == 'diagnosis: IPF')
pd2 = subset(pd2,characteristics_ch1.1 == 'diagnosis: IPF')
exp_idp = exp[,c(pd1$geo_accession,pd2$geo_accession)]пакетная коррекция
## 批次校正
BiocManager::install("sva")
library('sva')
## 批次信息
batch = data.frame(sample = c(pd1$geo_accession,pd2$geo_accession),
batch = c(pd1$platform_id,pd2$platform_id))PCA перед коррекцией партии
#install.packages('FactoMineR')
#install.packages('factoextra')
library("FactoMineR")
library("factoextra")
pca.plot = function(dat,col){
df.pca <- PCA(t(dat), graph = FALSE)
fviz_pca_ind(df.pca,
geom.ind = "point",
col.ind = col ,
addEllipses = TRUE,
legend.title = "Groups"
)
}
pca.plot(exp_idp,factor(batch$batch))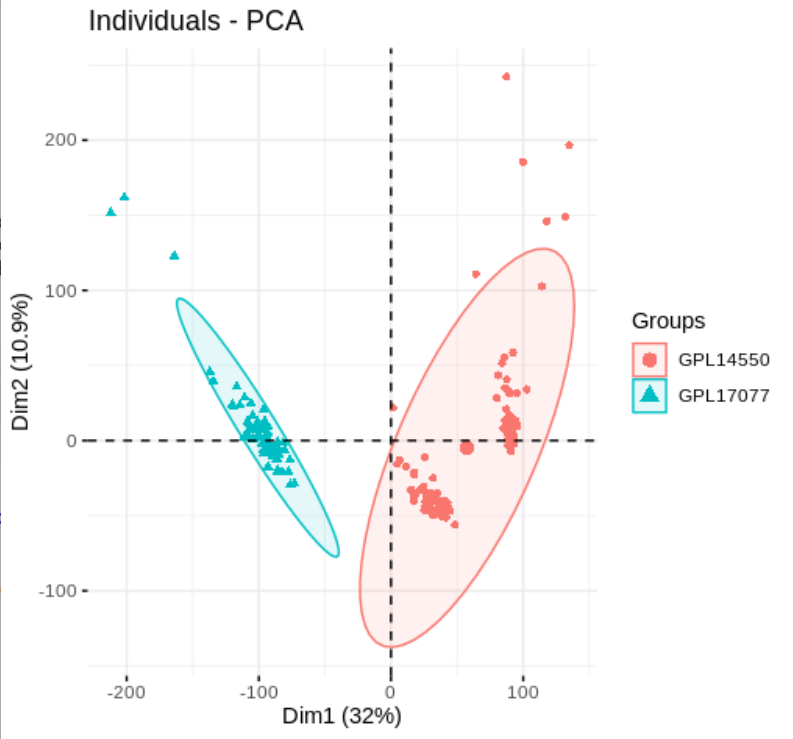
пакетная коррекция
## sva 批次校正
combat_exp <- ComBat(dat = as.matrix(log2(exp_idp+1)),
batch = batch$batch)
pca.plot(combat_exp,factor(batch$batch))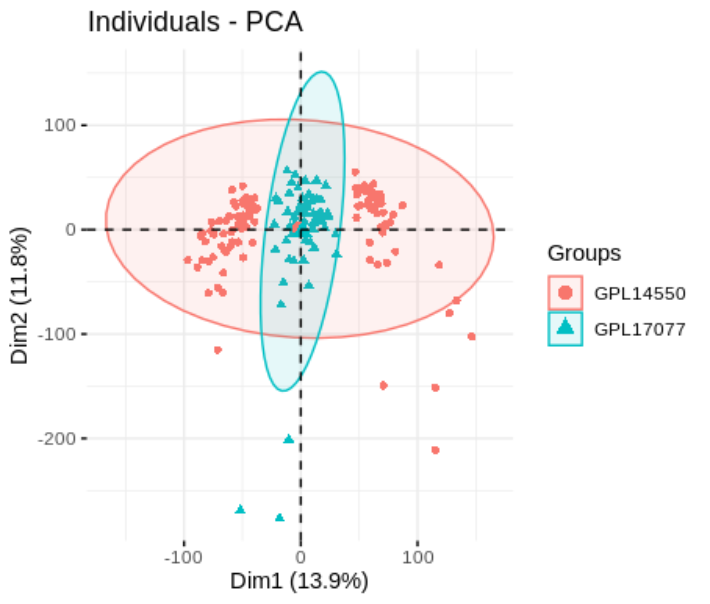
## 保存校正后的基因表达矩阵便于后续分析
save(combat_exp, eSet, file = "GSE70866.Rdata")Примечание: поскольку это только поверхностное воспроизведение, в процессе могут быть некоторые ошибки или недостатки.Я надеюсь, что каждый может критиковать и исправлять его.
Прошлый контент
Следуйте за Cell, чтобы научиться составлять карту | Расширенная версия карты вулкана
Подписывайтесь на Nat Commun, чтобы научиться рисовать | 2. Хронология графика
Подписывайтесь на Nat Commun, чтобы научиться рисовать | 4. Парный блок-график + анализ различий
