OS: Windows 10 x64
Python: Anaconda 4.4.0 (Python 2.7)
IDE: Spyder
Thanks Cameron Davidson-Pilon for his great work of Bayesian Methods for Hackers: Probabilistic Programming and Bayesian Inference.
下面我们来聊聊如何使用Python进行概率编程以及贝叶斯分析推断。
什么是概率编程?参考以下描述:
Another way of thinking about this: unlike a traditional program, which only runs in the forward directions, a probabilistic program is run in both the forward and backward direction. It runs forward to compute the consequences of the assumptions it contains about the world (i.e., the model space it represents), but it also runs backward from the data to constrain the possible explanations. In practice, many probabilistic programming systems will cleverly interleave these forward and backward operations to efficiently home in on the best explanations.
具体内涵大家可以不用太关心,实际它也只是编程。这里我们主要介绍Python模块PyMC的使用方法。
PyMC变量
父子变量
首先介绍PyMC变量中父变量和子变量。父变量会对其它变量产生影响,子变量不会,只会被影响,也就是父变量的作用变量。例如:
import pymc as pm
lambda_ = pm.Exponential("poisson_param",1)
data_generator = pm.Poisson("data_generator", lambda_)
data_plus_one = data_generator + 1
print lambda_.children
print data_generator.parents
print data_generator.children
print lambda_.value
print data_generator.value
print data_plus_one.value运行结果如下:
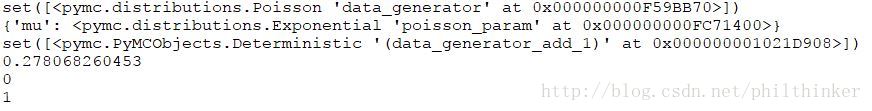
这样可以查看每个变量的父子关系,注意每个PyMC变量都有一个value属性,保存了当前的随机值。一个父变量可以有多个子变量,同样一个子变量也能有多个父变量。
随机与确定
随机变量是一种不确定的变量,即使获取了其所有父变量,它本身的值也是随机的。相反,确定变量在其父变量被确定后就不再随机。下面分开介绍:
随机变量
初始化:
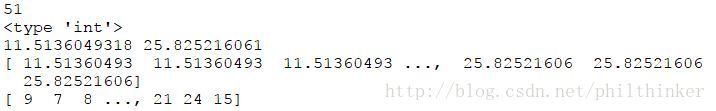
betas = pm.Uniform("betas", 0,1, size=10)
print betas.value
print type(betas)
print type(betas.value)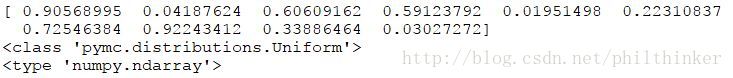
第一个参数是一个字符串,表示变量的名字。
产生新的随机值:
lambda_1 = pm.Exponential('lambda_1', 1)
lambda_2 = pm.Exponential('lambda_2', 1)
tau = pm.DiscreteUniform('tau', lower=0, upper=10)
print 'Initializing values ...'
print lambda_1.value
print lambda_2.value
print tau.value
lambda_1.random()
lambda_2.random()
tau.random()
print 'After calling random()'
print lambda_1.value
print lambda_2.value
print tau.value 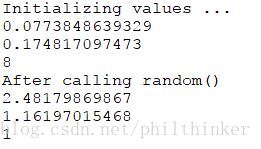
调用一次random()方法即可。
确定变量
申明一个确定变量的方法如下:
import numpy as np
N = 5
@pm.deterministic
def lambda_rep(tau=tau, lambda_1=lambda_1, lambda_2=lambda_2):
out = np.zeros(N)
out[:tau] = lambda_1 #Note that the local vars are values instead of stochastic vars
out[tau:] = lambda_2
return out
print type(lambda_rep)
print lambda_rep.value
print type(lambda_1+lambda_2)这里用到了Python装饰器,运行结果如下:

可以看出申明确定变量有两种方式:利用pm.deterministic装饰器或元素操作、加法、指数等类似方式。与随机变量不同,确定变量像标量或者NumPy那样传递,即传递的是变量的值。注意创建确定函数的时候,对函数中的每一个变量都要带关键字。
变量的观测值
PyMC随机变量都有一个observed参数,它取值为一个布尔值,默认为False。这个参数的作用是固定当前变量的取值。在创建变量的时候,需要赋予一个初值,即value参数,这个值即是希望加入的观测值,一般是一个NumPy数组。举例如下:
data = np.array([10 , 5 ])
fixed_variable = pm.Poisson("fxd" , 1 , value= data, observed= True )
print "value: " , fixed_variable. value
fixed_variable. random()
print "value after calling random(): " , fixed_variable. value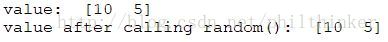
可以看出,调用random()方法前后随机变量的值并没有发生变化。
PyMC 随机数据产生
随机数据产生方法可以用来生成随机数据,举例如下:
tau2 = pm.rdiscrete_uniform(0,80)
print tau2
print type(tau2)
lambda2_1, lambda2_2 = pm.rexponential(0.05, 2)
print lambda2_1, lambda2_2
lambda_ = np.r_[lambda2_1*np.ones(tau2), lambda2_2*np.ones(80-tau2)]
print lambda_
data2 = pm.rpoisson(lambda_)
print data2PyMC建模方法
我们可以把所有创建的变量打包到 pm.Model 类中。有个这个类,我们可以一次性简便地分析变量。这是一个可选步骤。
model = pm. Model([fixed_variable, lambda_1, lambda_2, tau])在贝叶斯建模的时候,首先需要确定的是数据是如何产生的。给定一些观测数据后:
- 什么随机变量可以描述这些数据?假设我们选Poisson分布。
- 选定的分布有哪些参数?需要一个
λ 。 - 我们知道
λ 的具体值吗?如果不知道,对于λ 我们应该选一个怎样的分布?假设此时我们对λ 选择指数分布,它包含一个参数α 。 -
α 我们了解吗?如果不知道,我们可以继续为α 选择分布,但是未知已积累过多,此时最好停下来。我们可根据对于λ 的估计选择一个合适的α 。
一个简单的例子:贝叶斯A/B测试
"""
A/B Test
The key point locates in the issue that There is only ONE diversity between the two groups.
"""
import numpy as np
import pymc as pm
import matplotlib.pyplot as plt
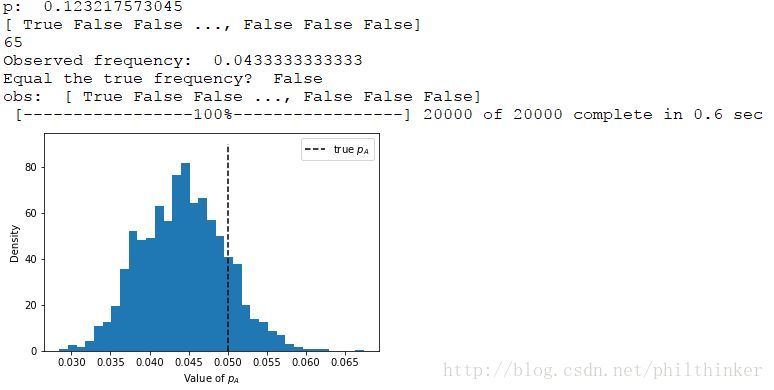
p = pm.Uniform('p', lower = 0, upper = 1)
print 'p: ',p.value
p_true = 0.05 # This is unkonwn in reality.
N = 1500
occurrences = pm.rbernoulli(p_true,N) # Each sample has a p_true chance of being 1.
print occurrences
print occurrences.sum()
print 'Observed frequency: ',occurrences.mean()
print 'Equal the true frequency? ',occurrences.mean()==p_true
obs = pm.Bernoulli('obs', p, value=occurrences, observed=True)
print 'obs: ',obs.value
mcmc = pm.MCMC([p,obs]) # Explained later
mcmc.sample(20000, 1000)
plt.vlines(p_true, 0, 90, linestyles='--', label='true $p_{A}$ ')
plt.hist(mcmc.trace('p')[:], bins=35, histtype='stepfilled', normed=True )
plt.xlabel('Value of $p_{A}$')
plt.ylabel('Density')
plt.legend()