基本概念
exemplar:聚类的中心点;
s(i,k):样本 i 和样本 k 之间的相似度,初始化时会有一个相似度矩阵;
preference:偏好参数,相似度矩阵中横轴纵轴索引相同的点,如s(i,i),表示数据点 i 作为聚类中心的程度。迭代开始前假设所有点成为聚类中心的能力相同,因此参考度一般设为相似度矩阵中所有值得最小值或者中位数,但是参考度越大则说明各数据点成为聚类中心的能力越强,则最终聚类中心的个数越多;
r(i,k): responsibility(吸引度),样本 k 适合作为样本 i 的聚类中心的累积信任度;
a(i,k): availability(归属度),样本 i 应该选择样本 k 作为其聚类中心的累积信任度;
damping factor:阻尼系数,为了避免 r(i,k) 和 a(i,k) 在更新时发生数值震荡;
算法流程
1、计算初始的相似度矩阵,将各点之间的吸引度 r(i,k) 和归属度 a(i,k) 初始化为 0;
2、更新各点之间的吸引度,随之更新各点之间的归属度,公式如下:
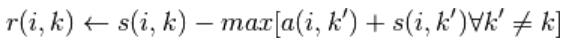
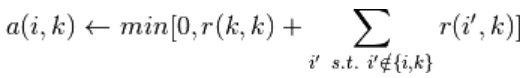
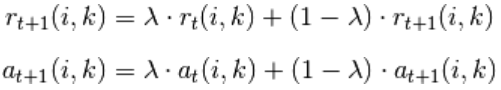
其中,
为阻尼系数;
3、确定当前样本 i 的代表样本(exemplar)点 k,k就是使{a(i,k)+r(i,k)}取得最大值的那个 k;
重复步骤 2 和步骤 3,直到所有的样本的所属都不再变化为止;
算法优缺点
优点:不需要提前规定聚类个数;
缺点:时间复杂度(
)和空间复杂度(
)都很高,其中 N 为样本个数,T 为迭代次数;
sklearn中的参数
[class sklearn.cluster.AffinityPropagation]
damping=0.5:阻尼系数,设置为 0.5 到 1 之间;
max_iter=200:最大迭代次数;
convergence_iter=15:聚类个数连续 convergence_iter 次迭代都不再改变,就停止迭代;
copy=True:在 scikit-learn 很多接口中都会有这个参数的,就是是否对输入数据进行 copy 操作,以便不修改用户的输入数据;
preference=None: array-like, shape (n_samples,) 或者 float,如果不指定,则都设为相似度矩阵中的中位数;
affinity=’euclidean’: string,“precomputed” 或者 “euclidean”,“euclidean” 采用负的欧几里得距离;
verbose=False: int 类型,是否输出详细信息;
示例代码
Demo of affinity propagation clustering algorithm
print(__doc__)
from sklearn.cluster import AffinityPropagation
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
# #############################################################################
# Generate sample data
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=300, centers=centers, cluster_std=0.5,
random_state=0)
# #############################################################################
# Compute Affinity Propagation
af = AffinityPropagation(preference=-50).fit(X)
cluster_centers_indices = af.cluster_centers_indices_
labels = af.labels_
n_clusters_ = len(cluster_centers_indices)
print('Estimated number of clusters: %d' % n_clusters_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels, metric='sqeuclidean'))
# #############################################################################
# Plot result
import matplotlib.pyplot as plt
from itertools import cycle
plt.close('all')
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
class_members = labels == k
cluster_center = X[cluster_centers_indices[k]]
plt.plot(X[class_members, 0], X[class_members, 1], col + '.')
plt.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
for x in X[class_members]:
plt.plot([cluster_center[0], x[0]], [cluster_center[1], x[1]], col)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()