R中的层次聚类
在本教程中,您将学习在 R 中对数据集执行层次聚类。更具体地说,您将了解:
介绍
顾名思义,聚类算法将一组数据点分组为子集或集群。这些算法的目标是创建内部一致但外部明显不同的集群。换句话说,集群中的实体应该尽可能相似,一个集群中的实体应该与另一个集群中的实体尽可能不同。
广义上讲,基于算法结构和操作的数据点聚类方法有两种,即凝聚法和分裂法。
- 凝聚法:凝聚法从不同(单一)集群中的每个观察开始,并连续将集群合并在一起,直到满足停止标准。
- 分裂:分裂方法从单个集群中的所有模式开始,并执行分裂直到满足停止标准。
在本教程中,您将关注凝聚法或自下而上的方法,在这种方法中,您将每个数据点作为自己的集群开始,然后根据某些相似性度量组合集群。这个想法也可以很容易地适用于分裂方法。
聚类之间的相似性通常是根据两个聚类之间的欧几里德距离等相异性度量来计算的。所以两个簇之间的距离越大越好。
您可以考虑许多距离度量来计算相异性度量,选择取决于数据集中的数据类型。例如,如果您的数据集中有连续的数值,您可以使用euclidean距离,如果数据是二进制的,您可以考虑Jaccard距离(当您在应用单热编码后处理分类数据进行聚类时很有帮助)。其他距离度量包括曼哈顿、明可夫斯基、堪培拉等。
聚类的预处理操作
在开始之前,您应该注意一些事项。
- 缩放
为了开始聚类过程,您必须标准化特征值的比例。这是因为每个观察的特征值都表示为 n 维空间中的坐标(n 是特征的数量),然后计算这些坐标之间的距离。如果这些坐标没有标准化,那么可能会导致错误的结果。
例如,假设您有三个人的身高和体重数据:A(6 英尺,75 公斤),B(6 英尺,77 公斤),C(8 英尺,75 公斤)。如果在二维坐标系中表示这些特征,身高和体重,并计算它们之间的欧几里德距离,则以下对之间的距离为:
AB : 2 台
交流电:2 台
好吧,距离度量表明 AB 和 AC 对是相似的,但实际上它们显然不是!AB 对比 AC 对更相似。因此,重要的是先缩放这些值,然后计算距离。
有多种方法可以对特征值进行归一化,您可以考虑通过应用以下转换对 [0,1] 之间的所有特征值 (x(i)) 的整个尺度进行标准化(称为最小-最大归一化):
x ( s ) = x ( i ) − m i n ( x ) / ( m a x ( x ) − m i n ( x ) )x(s)=x(i)−min(x)/(max(x)−min(x))
您可以normalize()为此使用 R 的函数,也可以编写自己的函数,例如:
standardize <- function(x){(x-min(x))/(max(x)-min(x))}
其他类型的缩放可以通过以下转换实现:
X (小号)= X (我)-米Ë一个Ñ (X )/小号d( x )x(s)=x(i)−mean(x)/sd(x)
其中 sd(x) 是特征值的标准偏差。这将确保您的特征值分布均值为 0,标准差为 1。您可以通过scale()R 中的函数实现这一点。
- 缺失值插补
事先处理数据集中的缺失/空/inf 值也很重要。有很多方法可以处理这些值,一种是删除它们,或者用均值、中值、众数来估算它们,或者使用一些高级回归技术。R有很多的包和函数来处理缺失值插补喜欢impute(),Amelia,Mice,Hmisc等你可以阅读Amelia这个教程。
层次聚类算法
层次凝聚聚类的关键操作是将两个最近的聚类重复组合成一个更大的聚类。首先需要回答三个关键问题:
- 你如何表示一个多点的集群?
- 你如何确定集群的“接近度”?
- 你什么时候停止合并集群?
希望在本教程结束时,您将能够回答所有这些问题。在应用层次聚类之前,让我们先看看它的工作原理:
- 它首先计算每对观察点之间的距离并将其存储在距离矩阵中。
- 然后它将每个点放在自己的集群中。
- 然后它开始根据与距离矩阵的距离合并最近的点对,结果簇的数量减少了 1。
- 然后它重新计算新集群和旧集群之间的距离,并将它们存储在新的距离矩阵中。
- 最后,它重复步骤 2 和 3,直到所有集群合并为一个集群。
有几种方法可以测量聚类之间的距离以决定聚类的规则,它们通常被称为链接方法。一些常见的链接方法是:
- Complete-linkage:在合并前计算集群之间的最大距离。
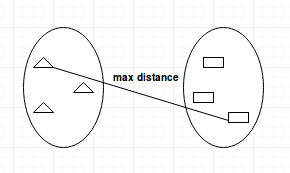
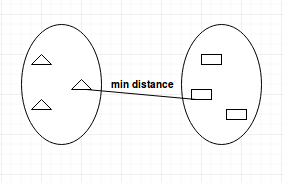
- Single-linkage:在合并之前计算集群之间的最小距离。此链接可用于检测数据集中的高值,这些值可能是异常值,因为它们将在最后合并。
- 平均链接:计算合并前集群之间的平均距离。

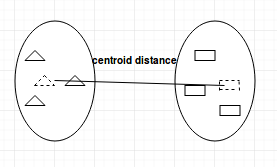
- Centroid-linkage:找到簇1的质心和簇2的质心,然后在合并前计算两者之间的距离。
联动方法的选择完全取决于您,没有一劳永逸的方法总能给您带来好的结果。不同的链接方法导致不同的集群。
树状图
在层次聚类中,您将对象分类为类似于树状图的层次结构,称为树状图。拆分或合并的距离(称为高度)显示在下面树状图的 y 轴上。
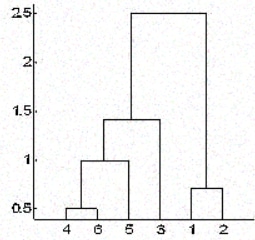
在上图中,首先将 4 和 6 合并为一个簇,例如簇 1,因为它们距离最近,然后是点 1 和点 2,例如簇 2。之后 5 合并到同一个簇 1 中,然后是3 导致两个集群。最后,两个集群合并为一个集群,这就是集群过程停止的地方。
现在可能让您感兴趣的一个问题是,您如何决定何时停止合并集群?嗯,这取决于您对数据的领域知识。例如,如果您根据他们在场地上的位置对场地上的足球运动员进行聚类,这将代表他们用于距离计算的坐标,那么您已经知道应该只以 2 个聚类结束,因为只能有两支球队在进行一场足球比赛.
但有时你也没有这些信息。在这种情况下,您可以利用树状图的结果来估算聚类数。您使用水平线切割树状图树,其高度使该线可以在不与合并点相交的情况下向上和向下遍历最大距离。在上述情况下,它将在高度 1.5 和 2.5 之间,如图所示:

如果您按如图所示进行切割,您最终只会得到两个簇。
请注意,不必只在这些地方进行切割,您可以根据需要的集群数量选择任意点作为切割点。例如,低于 1.5 和高于 1 的切割将为您提供 3 个集群。
请注意,这不是决定集群数量的硬性规则。您还可以考虑像剪影图、肘部图或一些数字度量(例如邓恩指数、休伯特伽马等)等图。它们显示误差随聚类数 (k) 的变化,您可以选择 k 的值,其中误差最小。
衡量集群的好坏
也许任何无监督学习任务中最重要的部分是对结果的分析。使用任何算法和任何参数集执行聚类后,您需要确保做得正确。但是你如何确定呢?
嗯,有很多措施可以做到这一点,也许最受欢迎的一种是Dunn's Index. 邓恩指数是最小簇间距离与最大簇内直径之间的比率。簇的直径是其两个最远点之间的距离。为了拥有分离良好且紧凑的集群,您应该瞄准更高的邓恩指数。
层次聚类在起作用
现在,您将应用所获得的知识来解决现实世界中的问题。
您将对seeds数据集应用层次聚类。该数据集包括对属于三种不同小麦品种的籽粒几何特性的测量:Kama、Rosa 和 Canadian。它具有描述种子特性的变量,如面积、周长、不对称系数等。每种小麦有 70 个观测值。您可以在此处找到有关数据集的详细信息。
首先使用该read.csv()函数将数据集导入到数据框中。
请注意,该文件没有任何标题并且以制表符分隔。为了保持结果的可重复性,您需要使用该set.seed()功能。
set.seed(786)
file_loc <- 'seeds.txt'
seeds_df <- read.csv(file_loc,sep = '\t',header = FALSE)
由于数据集没有任何列名称,您将根据数据描述自行指定列名称。
feature_name <- c('area','perimeter','compactness','length.of.kernel','width.of.kernal','asymmetry.coefficient','length.of.kernel.groove','type.of.seed')
colnames(seeds_df) <- feature_name
建议收集有关数据集的一些基本有用信息,例如其维度、数据类型和分布、NA 数量等。您将使用R 中的str(),summary()和is.na()函数来完成此操作。
str(seeds_df)
summary(seeds_df)
any(is.na(seeds_df))
请注意,此数据集的所有列都为数值。此数据集中没有您需要在聚类之前清理的缺失值。但是特征的尺度不同,你需要对其进行归一化。此外,数据已标记,并且您已经获得了关于哪个观测值属于哪种小麦品种的信息。
您现在将标签存储在单独的变量中,并type.of.seed从数据集中排除该列以进行聚类。稍后您将使用真实标签来检查您的聚类结果有多好。
seeds_label <- seeds_df$type.of.seed
seeds_df$type.of.seed <- NULL
str(seeds_df)
正如您会注意到的,您已经从数据集中删除了真正的标签列。
现在您将使用R'sscale()函数来缩放所有列值。
seeds_df_sc <- as.data.frame(scale(seeds_df))
summary(seeds_df_sc)
请注意,所有列的平均值为 0,标准差为 1。现在您已经对数据进行了预处理,是时候构建距离矩阵了。由于此处的所有值都是连续数值,因此您将使用euclidean距离方法。
dist_mat <- dist(seeds_df_sc, method = 'euclidean')
此时您应该决定要使用哪种链接方法并继续进行层次聚类。你可以尝试各种联动方式,然后再决定哪一种表现更好。在这里,您将继续使用average链接方法。
您将通过绘制将使用hclust(). 您可以通过method参数指定链接方法。
hclust_avg <- hclust(dist_mat, method = 'average')
plot(hclust_avg)
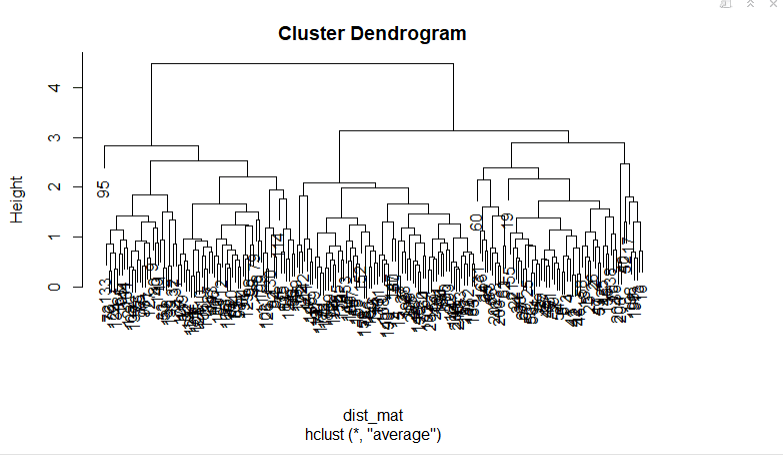
请注意树状图是如何构建的,每个数据点最终都合并到一个集群中,高度(距离)显示在 y 轴上。
接下来,您可以剪切树状图以创建所需数量的集群。由于在这种情况下您已经知道可能只有三种类型的小麦,因此您将选择簇数k = 3,或者如您在树状图中看到的那样,h = 3您会得到三个簇。您将使用 R 的cutree()函数以hclust_avg一个参数和另一个参数为h = 3or来切割树k = 3。
cut_avg <- cutree(hclust_avg, k = 3)
如果您想直观地看到树状图上的集群,您可以使用R的abline()函数绘制切割线并rect.hclust()使用如下代码所示的函数为树上的每个集群叠加矩形隔间:
plot(hclust_avg)
rect.hclust(hclust_avg , k = 3, border = 2:6)
abline(h = 3, col = 'red')
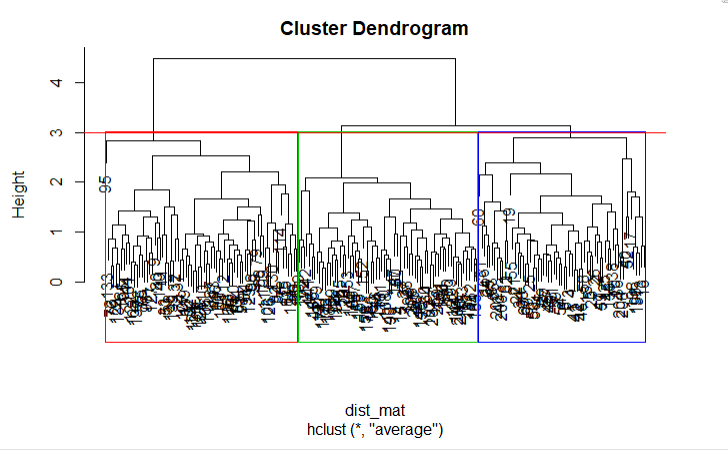
现在您可以看到三个不同颜色的盒子包围的三个集群。您还可以使用库中的color_branches()函数dendextend来可视化具有不同颜色分支的树。
请记住,您可以使用该install.packages('package_name', dependencies = TRUE)命令在 R 中安装软件包。
suppressPackageStartupMessages(library(dendextend))
avg_dend_obj <- as.dendrogram(hclust_avg)
avg_col_dend <- color_branches(avg_dend_obj, h = 3)
plot(avg_col_dend)
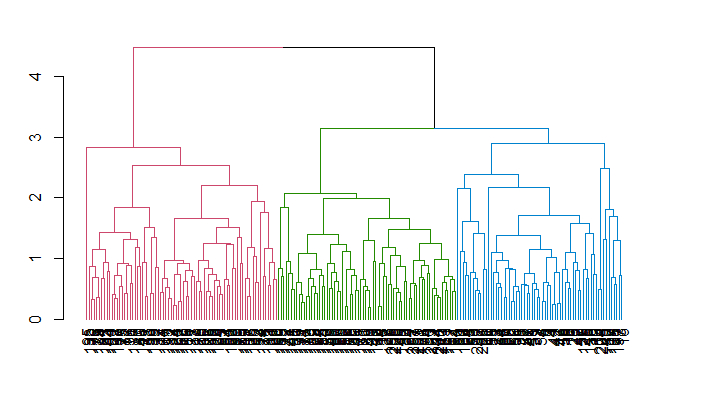
现在,您将从包中将获得的聚类结果附加回原始数据框中的列名clusterwith 下mutate(),dplyr并计算使用该count()函数为每个聚类分配了多少观测值。
suppressPackageStartupMessages(library(dplyr))
seeds_df_cl <- mutate(seeds_df, cluster = cut_avg)
count(seeds_df_cl,cluster)
您将能够看到在每个集群中分配了多少观测值。请注意,实际上根据标记数据,您对每种小麦都有 70 次观察。
通常根据您所做的聚类来评估两个特征之间的趋势,以便从数据聚类中提取更有用的见解。作为练习,您可以在 package.json 的帮助下分析小麦perimeter和area集群之间的趋势ggplot2。
suppressPackageStartupMessages(library(ggplot2))
ggplot(seeds_df_cl, aes(x=area, y = perimeter, color = factor(cluster))) + geom_point()
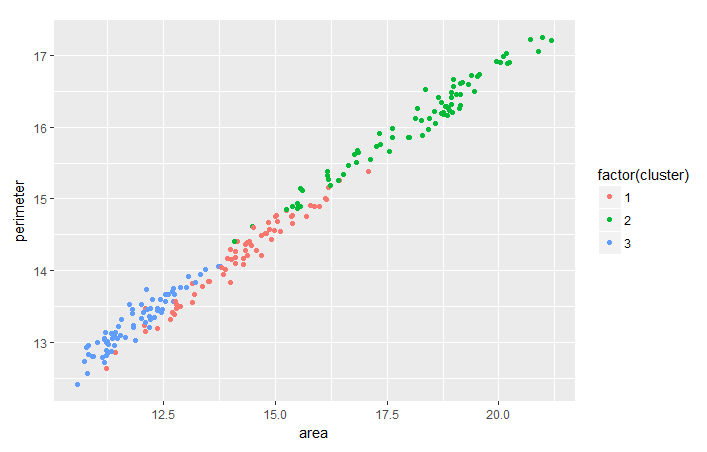
请注意,对于所有小麦品种,周长和面积之间似乎存在线性关系。
由于您已经拥有此数据集的真实标签,您还可以考虑使用该table()函数交叉检查您的聚类结果。
table(seeds_df_cl$cluster,seeds_label)
如果您查看生成的表格,您会清楚地看到包含 55 个或更多元素的三组。总体而言,您可以说您的聚类充分代表了不同类型的种子,因为最初您对每种小麦品种都有 70 次观察。较大的组代表集群和实际类型之间的对应关系。
请注意,在许多情况下,您实际上并没有真正的标签。在这些情况下,正如已经讨论过的,您可以采用其他措施,例如最大化邓恩指数。您可以使用库中的函数计算邓恩指数。此外,您可以考虑通过制作训练集和测试集来对结果进行交叉验证,就像您在任何其他机器学习算法中所做的那样,然后在您拥有真实标签时进行聚类。dunn()clValid
与K-Means聚类算法的比较
您可能听说过 k 均值聚类算法;如果没有,请查看本教程。两种算法之间存在许多根本区别,尽管在不同情况下,任何一种算法都可以比另一种算法表现得更好。其中一些差异是:
- 使用的距离:层次聚类实际上可以处理任何距离度量,而 k 均值依赖于欧几里德距离。
- 结果的稳定性:k-means 在初始化时需要一个随机步骤,如果重新运行该过程可能会产生不同的结果。在层次聚类中情况并非如此。
- 簇数:虽然您可以使用肘部图、剪影图等来计算 k 均值中正确的簇数,但分层也可以使用所有这些,但具有利用树状图的额外好处。
- 计算复杂性:K-means 的计算成本低于层次聚类,并且可以在合理的时间范围内在大型数据集上运行,这是 k-means 更受欢迎的主要原因。
结论
恭喜!您已完成本教程的学习。您学习了如何预处理数据、层次聚类的基础知识、距离度量和链接方法以及它在 R 中的用法。您还知道层次聚类与 k-means 算法的不同之处。做得好!但总有很多东西需要学习。我建议你看看我们的无监督学习 R课程。