Hoffmann H. Kernel PCA for novelty detection[J]. Pattern Recognition, 2007, 40(3): 863-874.
引
Novelty Detection: 给我的感觉有点像是奇异值检测,但是又不对,训练样本应该默认是好的样本。这个检测应该就是圈个范围,告诉我们在这个范围里的数据是这个类的,外面的不是这个类的,所以论文里也称之为:one-class classification
论文就是利用kernel PCA来寻找这么一个边界(虽然我不知道这个边界是怎么获得的),并讨论了kernel的参数以及载荷向量个数q的选取。

作者给出了几种方法的比较(SVM及其变种),说明了利用reconstruction error设定边界的优势。
主要内容
大部分符合和kernel PCA的符号是一致的,这里就不多赘述了,先给出reconstruction error的定义:
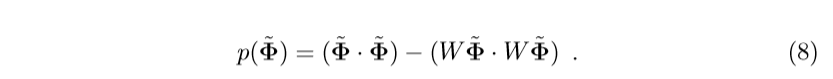
其中
的行向量是特征向量(高维空间中的,这里只是如此表示,就像kernel PCA精髓,不必显示地表示出),
是中心化后的
。
很显然,上面的式子的第二项实际上就是
在子空间中投影的部分的内积,所以
就是重构的被舍弃的误差。很自然,同一类数据
的值应该相对比较大。所以
选择的特征向量是主特征向量(对应的特征值比较大)。
注:可以发现
这个式子如何计算呢?
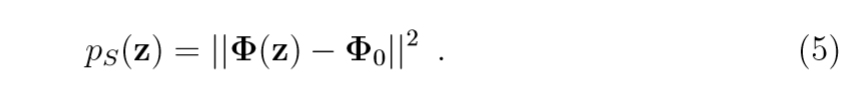
其中 ,于是, 可以用下式来计算:
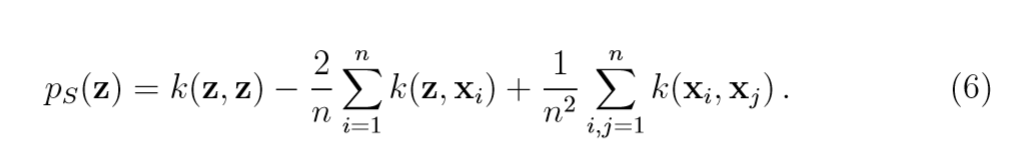
注: 第二项与Parzen Windows有很大关联
令:

其中
为第
个特征向量。
易得:
所以:
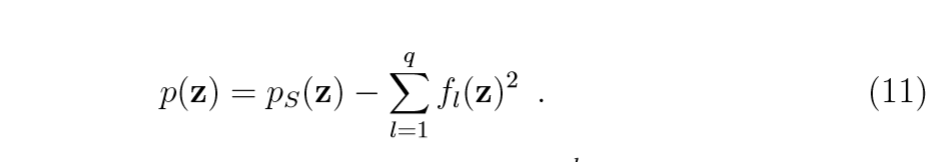
需要注意的是(6)和(9)中有许多重复计算的地方,可以先计算这些部分再进行整理会简单很多。
注:kernel为高斯核的时候, 的值是固定,可以理解为高维空间中向量的模大小一致,个人认为这是非常好的一个性质,这也是在数值实验中,高斯核的性能远远超出多项式核的原因(至少在这个方面是如此的)。
的选择
高斯核的形态为:
当
的时候,
,这时
彼此正交,所以
不应当太小。
当
的时候:
可以证明,此时的reconstruction error的效用采用
和采用
的类似的。
首先:
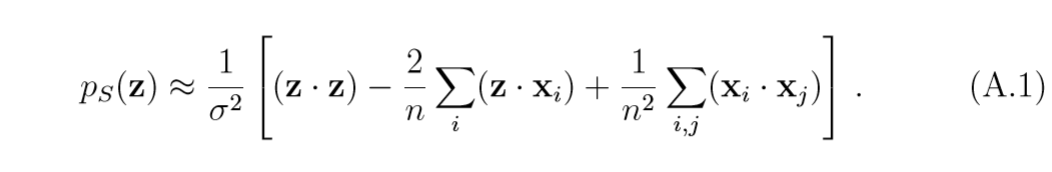

均只差一个比例常数
,又:

也差一个比例常数
,所以此时采用
和
并没有太大的差别。这意味着,
不能太大。
最后再提一下 的选择,显然 应当足够大,这个参数可以通过特征值来选择。
数值实验
数值实验采用的kernel均为高斯核,多项式核效果核论文中的差别有点大就不提了。
矩形框
数据是如此分布的(只是框定了范围,里面的数据点是从均匀分布中选取的):0.4 ~ 0.6, 2.3 ~ 2.6

q = 20,

q = 20,

q = 30

q = 30

q = 40

q = 40

q = 80

q=80

下面是q=50.
的error在y=1.5处左右的值

spiral
原始数据如下(只是这个形状的):

q = 40

q = 40

下面是q=40,
的
左右处的error值:

代码
import numpy as np
class KernelPCA:
def __init__(self, data, kernel=2, pra=0.4):
self.__n, self.__d = data.shape
self.__data = np.array(data, dtype=float)
self.kernel = self.kernels(kernel, pra)
self.__K = self.center()
@property
def shape(self):
return self.__n, self.__d
@property
def data(self):
return self.data
@property
def K(self):
return self.__K
@property
def alpha(self):
return self.__alpha
@property
def eigenvalue(self):
return self.__value
def kernels(self, label, pra):
"""
数据是一维的时候可能有Bug
:param label: 1:多项式;2:exp
:param pra:1: d; 2: sigma
:return: 函数 或报错
"""
if label is 1:
return lambda x, y: (x @ y) ** pra
elif label is 2:
return lambda x, y: \
np.exp(-(x-y) @ (x-y) / (2 * pra ** 2))
else:
raise TypeError("No such kernel...")
def center(self):
"""中心化"""
oldK = np.zeros((self.__n, self.__n), dtype=float)
one_n = np.ones((self.__n, self.__n), dtype=float)
for i in range(self.__n):
for j in range(i, self.__n):
x = self.__data[i]
y = self.__data[j]
oldK[i, j] = oldK[j, i] = self.kernel(x, y)
return oldK - 2 * one_n @ oldK + one_n @ oldK @ one_n
def ordereig(self, A):
"""晕了,没想到linalg.eig出来的特征值不一定是按序的"""
value, vector = np.linalg.eig(A)
order = np.argsort(value)[::-1]
value = value[order]
vector = vector.T[order].T
return value, vector
def processing(self):
"""实际上就是K的特征分解,再对alpha的大小进行一下调整"""
value, alpha = self.ordereig(self.__K)
index = value > 0
value = value[index]
alpha = alpha[:, index] * (1 / np.sqrt(value))
self.__alpha = alpha
self.__value = value / self.__n
def score(self, x):
"""来了一个新的样本,我们进行得分"""
k = np.zeros(self.__n)
for i in range(self.__n):
y = self.__data[i]
k[i] = self.kernel(x, y)
return k @ self.__alpha
class NoveltyDetection:
def __init__(self, data, q, kernel=2, pra=0.4):
self.kernel = self.kernels(kernel, pra) #选择kernel
kernelPCA = KernelPCA(data, kernel, pra) #进行kernel PCA
kernelPCA.processing() #进行kernel PCA
self.__alpha = kernelPCA.alpha[:, :q] #获取相应的alpha
self.__K = kernelPCA.K #获取K
self.basedata() #用于计算reconstruction error
self.__data = np.array(data, dtype=float)
self.__shape = kernelPCA.shape
del kernelPCA #我不知道这么做是否有意义
@property
def alpha(self):
return self.__alpha
@property
def K(self):
return self.__K
@property
def data(self):
return self.__data
@property
def shape(self):
return self.__shape
def kernels(self, label, pra):
"""
数据是一维的时候可能有Bug
:param label: 1:多项式;2:exp
:param pra:1: d; 2: sigma
:return: 函数 或报错
"""
if label is 1:
return lambda x, y: (x @ y) ** pra
elif label is 2:
return lambda x, y: \
np.exp(-(x-y) @ (x-y) / (2 * pra ** 2))
else:
raise TypeError("No such kernel...")
def basedata(self):
self.unitmean = np.mean(self.K, 1)
self.allmean = np.mean(self.K)
def detection(self, z):
n = self.shape[0]
phiz = np.zeros(n)
for i in range(n):
phiz[i] = self.kernel(z, self.data[i])
psz = self.kernel(z, z) - np.mean(phiz) * 2 + \
self.allmean
temp1 = -np.mean(phiz) + self.allmean
temp2 = np.array([
phiz[i] - self.unitmean[i] + temp1
for i in range(n)
], dtype=float)
pz = psz - np.sum((self.alpha.T @ temp2) ** 2)
return pz
"""
import matplotlib.pyplot as plt
x = (np.random.rand(1000) + 0.4 / 2.2) * 2.2
y = (np.random.rand(1000) + 0.4 / 2.2) * 2.2
func_and = lambda x: x[0] and x[1]
func_or = lambda x: x[0] or x[1]
def findindex(x, x1, x2, x3, x4):
index1 = zip(x > x1, x < x2)
index2 = zip(x > x3, x < x4)
index3 = list(map(func_and, index1))
index4 = list(map(func_and, index2))
index = list(map(func_or, zip(index3, index4)))
return index
xindex = findindex(x, 0.4, 0.7, 2.3, 2.6)
yindex = findindex(y, 0.4, 0.7, 2.3, 2.6)
index = list(map(func_or, zip(xindex, yindex)))
x = x[index]
y = y[index]
data = np.column_stack((x, y))
test = NoveltyDetection(data, 40, kernel=2, pra=0.2)
Y, X = np.mgrid[0:3:100j, 0:3:100j]
newdata = np.zeros(X.shape)
for i in range(X.shape[0]):
for j in range(X.shape[1]):
z = np.array([X[i, j], Y[i, j]])
newdata[i, j] = test.detection(z)
plt.scatter(X, Y, c=newdata)
plt.xlim(0, 3)
plt.ylim(0, 3)
plt.show()
"""