简单介绍:
k-means聚类属于无监督学习的一种,在没有给与labels的情况下,将数据分成指定的K类。
它将相似的对象归到一个簇中,将不相似的对象归到不同簇中,相似这一概念,取决于所选择的相似度计算方法。
K-means是发现给定数据集的K个簇的聚类算法,之所以称之为K均值,是因为他可以发现K个不同的簇,且每个簇的中心采用簇中所含值得均值计算而成。
簇的个数是用户指定的,每一个簇通过其质心,即簇中所有点的中心来描述。
聚类于分类算法最大的区别在于,分类的目标类别已知,但是聚类目标类别是未知的。
思想:
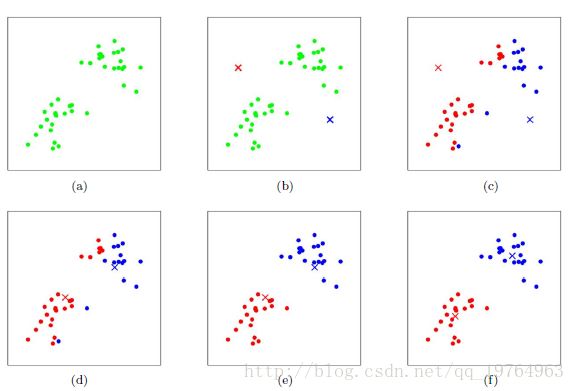
这次mark和上一次不一样吧?那说明这次细分有效果了
接下来,进行第二次站队,也就是所有的点再一次计算和两个mark的距离,认领新的mark归属,好了,新的局势再次形成,接下来继续选择合适的mark新位置,和上次对比发现,依然后变化,恩!那就对了,不断重复这两个步骤,直到mark位置几乎不变,那就完成了聚类过程。

1.载入数据
def loadDataSet(fileName):
'''
加载数据集
:param fileName:
:return:
'''
# 初始化一个空列表
dataSet = []
# 读取文件
fr = open(fileName)
# 循环遍历文件所有行
for line in fr.readlines():
# 切割每一行的数据
curLine = line.strip().split('\t')
# 将数据转换为浮点类型,便于后面的计算
# fltLine = [float(x) for x in curLine]
# 将数据追加到dataMat
fltLine = list(map(float,curLine)) # 映射所有的元素为 float(浮点数)类型
dataSet.append(fltLine)
# 返回dataMat
return mat(dataSet)
2.求向量距离
K均值聚类中需要计算数据和质心的距离,常见的距离有欧氏距离(Euclidean distance)和曼哈顿距离(Manhattan distance),本处采用欧式距离。def distEclud(vecA, vecB):
'''
欧氏距离计算函数
:param vecA:
:param vecB:
:return:
'''
return sqrt(sum(power(vecA - vecB, 2)))
3.随机生成k个点作为初始质心
def randCent(dataMat, k):
'''
为给定数据集构建一个包含K个随机质心的集合,
随机质心必须要在整个数据集的边界之内,这可以通过找到数据集每一维的最小和最大值来完成
然后生成0到1.0之间的随机数并通过取值范围和最小值,以便确保随机点在数据的边界之内
:param dataMat:
:param k:
:return:
'''
# 获取样本数与特征值
m, n = dataMat.shape
# 初始化质心,创建(k,n)个以零填充的矩阵
centroids = mat(zeros((k, n)))
# 循环遍历特征值
for j in range(n):
# 计算每一列的最小值
minJ = min(dataMat[:, j])
# 计算每一列的范围值
rangeJ = float(max(dataMat[:, j]) - minJ)
# 计算每一列的质心,并将值赋给centroids
centroids[:, j] = mat(minJ + rangeJ * random.rand(k, 1))
# 返回质心
return centroids
4.K均值聚类算法实现

def kMeans(dataMat, k, distMeas=distEclud, createCent=randCent):
'''
创建K个质心,然后将每个店分配到最近的质心,再重新计算质心。
这个过程重复数次,直到数据点的簇分配结果不再改变为止
:param dataMat: 数据集
:param k: 簇的数目
:param distMeans: 计算距离
:param createCent: 创建初始质心
:return:
'''
# 获取样本数和特征数
m, n = dataMat.shape
# 初始化一个矩阵来存储每个点的簇分配结果
# clusterAssment包含两个列:一列记录簇索引值,
# 第二列存储误差(误差是指当前点到簇质心的距离,后面会使用该误差来评价聚类的效果)
clusterAssment = mat(zeros((m, 2)))
# 创建质心,随机K个质心
centroids = createCent(dataMat, k)
# 初始化标志变量,用于判断迭代是否继续,如果True,则继续迭代
clusterChanged = True
while clusterChanged:
clusterChanged = False
# 遍历所有数据找到距离每个点最近的质心,
# 可以通过对每个点遍历所有质心并计算点到每个质心的距离来完成
for i in range(m):
minDist = inf
minIndex = -1
for j in range(k):
# 计算数据点到质心的距离
# 计算距离是使用distMeas参数给出的距离公式,默认距离函数是distEclud
distJI = distMeas(centroids[j, :], dataMat[i, :])
# 如果距离比minDist(最小距离)还小,更新minDist(最小距离)和最小质心的index(索引)
if distJI < minDist:
minDist = distJI
minIndex = j
# 如果任一点的簇分配结果发生改变,则更新clusterChanged标志
if clusterAssment[i, 0] != minIndex: clusterChanged = True
# 更新簇分配结果为最小质心的index(索引),minDist(最小距离)的平方
clusterAssment[i, :] = minIndex, minDist ** 2
# print(centroids)
# 遍历所有质心并更新它们的取值
for cent in range(k):
# 通过数据过滤来获得给定簇的所有点
ptsInClust = dataMat[nonzero(clusterAssment[:, 0].A == cent)[0]]
# 计算所有点的均值,axis=0表示沿矩阵的列方向进行均值计算
centroids[cent, :] = mean(ptsInClust, axis=0)
# 返回所有的类质心与点分配结果
return centroids, clusterAssment
5.数据可视化
def showCluster(dataSet, k, clusterAssment, centroids):
fig = plt.figure()
plt.title("K-means")
ax = fig.add_subplot(111)
data = []
for cent in range(k): #提取出每个簇的数据
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]] #获得属于cent簇的数据
data.append(ptsInClust)
for cent, c, marker in zip( range(k), ['r', 'g', 'b', 'y'], ['^', 'o', '*', 's'] ): #画出数据点散点图
ax.scatter(data[cent][:, 0], data[cent][:, 1], s=80, c=c, marker=marker)
ax.scatter(np.array(centroids)[:, 0], np.array(centroids)[:, 1], s=1000, c='black', marker='+', alpha=1) #画出质心点
ax.set_xlabel('X Label')
ax.set_ylabel('Y Label')
plt.show()




一些说明:
- 每次迭代中都要对每一个簇进行划分,在其中选择最大程度降低误差平方和簇进行聚类
- 当使用KMeans()函数且指定的聚类数目为2时,会得到编号为0和1的两个簇,将编号为0的簇编号改为输入簇的编号,编号为1的簇编号改为所有簇的数目len(centList),即在原先簇上追加一个簇
def biKmeans(dataMat, k, distMeas=distEclud):
'''
在给定数据集,所期望的簇数目和距离计算方法的条件下,函数返回聚类结果
:param dataMat:
:param k:
:param distMeas:
:return:
'''
m, n = dataMat.shape
# 创建一个矩阵来存储数据集中每个点的簇分配结果及平方误差
clusterAssment = mat(zeros((m, 2)))
# 计算整个数据集的质心,并使用一个列表来保留所有的质心
centroid0 = mean(dataMat, axis=0).tolist()[0]
centList = [centroid0]
# 遍历数据集中所有点来计算每个点到质心的误差值
for j in range(m):
clusterAssment[j, 1] = distMeas(mat(centroid0), dataMat[j, :]) ** 2
# 对簇不停的进行划分,直到得到想要的簇数目为止
while (len(centList) < k):
# 初始化最小SSE为无穷大,用于比较划分前后的SSE
lowestSSE = inf
# 通过考察簇列表中的值来获得当前簇的数目,遍历所有的簇来决定最佳的簇进行划分
for i in range(len(centList)):
# 对每一个簇,将该簇中的所有点堪称一个小的数据集
ptsInCurrCluster = dataMat[nonzero(clusterAssment[:, 0].A == i)[0], :]
# 将ptsInCurrCluster输入到函数kMeans中进行处理,k=2,
# kMeans会生成两个质心(簇),同时给出每个簇的误差值
centroidMat, splitClustAss = kMeans(ptsInCurrCluster, 2, distMeas)
# 将误差值与剩余数据集的误差之和作为本次划分的误差
sseSplit = sum(splitClustAss[:, 1])
sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:, 0].A != i)[0], 1])
print('sseSplit, and notSplit: ', sseSplit, sseNotSplit)
# 如果本次划分的SSE值最小,则本次划分被保存
if (sseSplit + sseNotSplit) < lowestSSE:
bestCentToSplit = i
bestNewCents = centroidMat
bestClustAss = splitClustAss.copy()
lowestSSE = sseSplit + sseNotSplit
# 找出最好的簇分配结果
# 调用kmeans函数并且指定簇数为2时,会得到两个编号分别为0和1的结果簇
bestClustAss[nonzero(bestClustAss[:, 0].A == 1)[0], 0] = len(centList)
# 更新为最佳质心
bestClustAss[nonzero(bestClustAss[:, 0].A == 0)[0], 0] = bestCentToSplit
print('the bestCentToSplit is: ', bestCentToSplit)
print('the len of bestClustAss is: ', len(bestClustAss))
# 更新质心列表
# 更新原质心list中的第i个质心为使用二分kMeans后bestNewCents的第一个质心
centList[bestCentToSplit] = bestNewCents[0, :].tolist()[0]
# 添加bestNewCents的第二个质心
centList.append(bestNewCents[1, :].tolist()[0])
# 重新分配最好簇下的数据(质心)以及SSE
clusterAssment[nonzero(clusterAssment[:, 0].A == bestCentToSplit)[0], :] = bestClustAss
return mat(centList), clusterAssment
二分K均值之所以稳定,是由于初始质心不再是随机生成K个,而是基于全部数据的平均值先生成一个质心,然后基于最优的方法分裂成2个质心,然后再对现有的2个判断下哪个簇的误差较大,将其再分裂成2个。如此n+1+1+1每次优化分裂一个簇,直到达到k个簇结束优化。
机器学习实战中最后给了一个案例介绍聚类的一个应用场景,小伙伴们要出去游玩,选择了几个地方,想打车到几个地方的中心点后步行前往,给出最优路线。
采用二分K-均值法本身不难,我却被其中另一个问题给考倒了,因为获得的地点是经纬度坐标,如何求地球妈妈球面积上坐标之间的距离?
https://www.cnblogs.com/softfair/p/distance_of_two_latitude_and_longitude_points.html
这个小伙伴讲的很透彻了,基本是就是利用平面的问题去求解立体的问题,原理图如下:
代码中需要添加一个求坐标距离的辅助函数:
def distSLC(vecA, vecB):
'''
返回地球表面两点间的距离,单位是英里
给定两个点的经纬度,可以使用球面余弦定理来计算亮点的距离
:param vecA:
:param vecB:
:return:
'''
# 经度和维度用角度作为单位,但是sin()和cos()以弧度为输入.
# 可以将江都除以180度然后再诚意圆周率pi转换为弧度
a = sin(vecA[0, 1] * pi / 180) * sin(vecB[0, 1] * pi / 180)
b = cos(vecA[0, 1] * pi / 180) * cos(vecB[0, 1] * pi / 180) * cos(pi * (vecB[0, 0] - vecA[0, 0]) / 180)
return arccos(a + b) * 6371.0
利用二分K-均值求质心,然后将结果展示到地图上:
def clusterClubs(fileName, imgName, numClust=5):
'''
将文本文件的解析,聚类以及画图都封装在一起
:param fileName: 文本数据路径
:param imgName: 图片路径
:param numClust: 希望得到的簇数目
:return:
'''
# 创建一个空列表
datList = []
# 打开文本文件获取第4列和第5列,这两列分别对应维度和经度,然后将这些值封装到datList
for line in open(fileName).readlines():
lineArr = line.split('\t')
datList.append([float(lineArr[4]), float(lineArr[3])])
datMat = mat(datList)
# 调用biKmeans并使用distSLC函数作为聚类中使用的距离计算方式
myCentroids, clustAssing = biKmeans(datMat, numClust, distMeas=distSLC)
# 创建一幅图和一个举行,使用该矩形来决定绘制图的哪一部分
fig = plt.figure()
rect = [0.1, 0.1, 0.8, 0.8]
# 构建一个标记形状的列表用于绘制散点图
scatterMarkers = ['s', 'o', '^', '8', 'p', 'd', 'v', 'h', '>', '<']
axprops = dict(xticks=[], yticks=[])
ax0 = fig.add_axes(rect, label='ax0', **axprops)
# 使用imread函数基于一幅图像来创建矩阵
imgP = plt.imread(imgName)
# 使用imshow绘制该矩阵
ax0.imshow(imgP)
# 再同一幅图上绘制一张新图,允许使用两套坐标系统并不做任何缩放或偏移
ax1 = fig.add_axes(rect, label='ax1', frameon=False)
# 遍历每一个簇并将它们一一画出来,标记类型从前面创建的scatterMarkers列表中得到
for i in range(numClust):
ptsInCurrCluster = datMat[nonzero(clustAssing[:, 0].A == i)[0], :]
# 使用索引i % len(scatterMarkers)来选择标记形状,这意味这当有更多簇时,可以循环使用这标记
markerStyle = scatterMarkers[i % len(scatterMarkers)]
# 使用十字标记来表示簇中心并在图中显示
ax1.scatter(ptsInCurrCluster[:, 0].flatten().A[0], ptsInCurrCluster[:, 1].flatten().A[0], marker=markerStyle,
s=90)
ax1.scatter(myCentroids[:, 0].flatten().A[0], myCentroids[:, 1].flatten().A[0], marker='+', s=300)
plt.show()
fileName='./kmeans/places.txt'
imgName='./kmeans/Portland.png'
clusterClubs(fileName, imgName, numClust=5)


sklearn.cluster.k_means
-
sklearn.cluster.k_means( X, n_clusters, init='k-means++', precompute_distances='auto', n_init=10, max_iter=300, verbose=False, tol=0.0001, random_state=None, copy_x=True, n_jobs=1, algorithm='auto', return_n_iter=False )
-
Parameters: X : array-like or sparse matrix, shape (n_samples, n_features)
The observations to cluster.
n_clusters : int
The number of clusters to form as well as the number of centroids to generate.
init : {‘k-means++’, ‘random’, or ndarray, or a callable}, optional
Method for initialization, default to ‘k-means++’:
‘k-means++’ : selects initial cluster centers for k-mean clustering in a smart way to speed up convergence. See section Notes in k_init for more details.
‘random’: generate k centroids from a Gaussian with mean and variance estimated from the data.
If an ndarray is passed, it should be of shape (n_clusters, n_features) and gives the initial centers.
If a callable is passed, it should take arguments X, k and and a random state and return an initialization.
precompute_distances : {‘auto’, True, False}
Precompute distances (faster but takes more memory).
‘auto’ : do not precompute distances if n_samples * n_clusters > 12 million. This corresponds to about 100MB overhead per job using double precision.
True : always precompute distances
False : never precompute distances
n_init : int, optional, default: 10
Number of time the k-means algorithm will be run with different centroid seeds. The final results will be the best output of n_init consecutive runs in terms of inertia.
max_iter : int, optional, default 300
Maximum number of iterations of the k-means algorithm to run.
verbose : boolean, optional
Verbosity mode.
tol : float, optional
The relative increment in the results before declaring convergence.
random_state : int, RandomState instance or None, optional, default: None
If int, random_state is the seed used by the random number generator; If RandomState instance, random_state is the random number generator; If None, the random number generator is the RandomState instance used by np.random.
copy_x : boolean, optional
When pre-computing distances it is more numerically accurate to center the data first. If copy_x is True, then the original data is not modified. If False, the original data is modified, and put back before the function returns, but small numerical differences may be introduced by subtracting and then adding the data mean.
n_jobs : int
The number of jobs to use for the computation. This works by computing each of the n_init runs in parallel.
If -1 all CPUs are used. If 1 is given, no parallel computing code is used at all, which is useful for debugging. For n_jobs below -1, (n_cpus + 1 + n_jobs) are used. Thus for n_jobs = -2, all CPUs but one are used.
algorithm : “auto”, “full” or “elkan”, default=”auto”
K-means algorithm to use. The classical EM-style algorithm is “full”. The “elkan” variation is more efficient by using the triangle inequality, but currently doesn’t support sparse data. “auto” chooses “elkan” for dense data and “full” for sparse data.
return_n_iter : bool, optional
Whether or not to return the number of iterations.
Returns: centroid : float ndarray with shape (k, n_features)
Centroids found at the last iteration of k-means.
label : integer ndarray with shape (n_samples,)
label[i] is the code or index of the centroid the i’th observation is closest to.
inertia : float
The final value of the inertia criterion (sum of squared distances to the closest centroid for all observations in the training set).
best_n_iter : int
Number of iterations corresponding to the best results. Returned only if return_n_iter is set to True.
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
# 加载数据集
dataMat = []
fr = open("E:\python_code\Python_algorithm\ml\MachineLearning-master\input/10.KMeans/testSet.txt") # 注意,这个是相对路径,请保证是在 MachineLearning 这个目录下执行。
for line in fr.readlines():
curLine = line.strip().split('\t')
fltLine = list(map(float,curLine)) # 映射所有的元素为 float(浮点数)类型
dataMat.append(fltLine)
# 训练模型
km = KMeans(n_clusters=4) # 初始化
km.fit(dataMat) # 拟合
km_pred = km.predict(dataMat) # 预测
print(km_pred)
centers = km.cluster_centers_ # 质心
# 可视化结果
plt.scatter(np.array(dataMat)[:, 1], np.array(dataMat)[:, 0], c=km_pred)
plt.scatter(centers[:, 1], centers[:, 0], c="r")
plt.show()




