Two-Stage Approach for Segmenting Gross Tumor Volume in Head and Neck Cancer with CT and PET Imaging
头颈部肿瘤CT和PET影像二期分割方法
Abstract
头颈部癌症的放射治疗计划包括在CT图像上仔细描绘肿瘤目标体积,通常还需要PET扫描的帮助。在这项研究中,作为HECKTOR挑战的一部分,我们描述了一种使用深卷积神经网络的两阶段方法自动分割原发肿瘤的大体肿瘤体积的方法。我们训练一个分类网络来选择可能包含肿瘤的轴向切片,然后将这些切片输入分割网络,生成二值分割图。在由53名患者组成的测试集上,我们获得了平均骰子相似系数为0.644,平均准确率为0.694,平均召回率为0.667。
Introduction
在这篇文章中,我们描述了我们团队应对这一挑战的方法。我们将改进的ResNet和改进的U-Net组合起来,形成了一个两级深度神经网络,用于肿瘤分割。用改进的ResNet进行分类,选择可能含有GTVt的轴状切片,用改进的U-Net在先前网络选择的轴状切片中分割GTVt。
Materials
Dataset Description
训练数据集包括来自四家医院的201名患者。对于每个患者,输入由共同配准的三维CT图像(HU)和FDG-PET图像(标准摄取值,SUV)组成,标签是基于CT分辨率的GTVt的三维二进制分布(0和1)。体素大小因患者不同而不同,因为他们来自不同的机构。体素大小在x和y方向上约为1.0 mm,但在z方向上从1.5 mm到3.0 mm不等。提供144 mmx144 mmx144 mm的包围盒,指示包含口咽肿瘤的区域。
值得注意的是,由于PET图像上的SUV指示代谢活动量,预计SUV升高的区域包括原发肿瘤、肿瘤相关淋巴结和其他正常组织/器官,这些组织/器官具有正常的高代谢活动,但这一挑战的目标是单独分割原发肿瘤(GTVt),同时排除肿瘤相关淋巴结(GTVn)。测试数据集由来自一家医院的53名患者组成。为每个患者提供的输入数据类似于训练数据集,并且不提供分割掩码。
Data Preprocessing
将原始的三维CT和PET图像配准,并用双线性插值法重新采样到1 mm、1 mm、1 mm像素间距。然后我们裁剪了144 mmx144 mmx144 mm的各向同性体积,它在x和y方向上随机移动了10 mm,用于数据增强,以避免在训练期间模型过拟合。
CT体积被削减到[−150,150]Hu范围,这是头颈部区域的常见设置,其中感兴趣的组织主要是软组织、液体和脂肪[1]。此外,通过划分裁剪体积内所有体素的HUs的标准差来进一步标准化每个CT体积,以最小化扫描仪间的影响。受以前研究中使用的预处理方法的启发[3],每个PET体积被修剪到[0.2*SUVmax,10.0]SUV范围,然后划分10.0以保持CT和PET体积值之间的数量级相似。(这一部分不是很明白,以后可以仔细研究一下)
然后,我们将CT和PET体积切片成2D轴向切片,并将相应的CT和PET切片连接成2个通道,这样,对于每个患者,有144个2D切片大小(144,144,2)。
Methods
Overall Network Scheme
我们使用了两个阶段的方法,分别训练一个分类CNN和一个分割U-Net。分类网络选择一系列包含GTVt的2D切片,然后分割网络基于这些切片预测分割图。总体方案如图1所示。
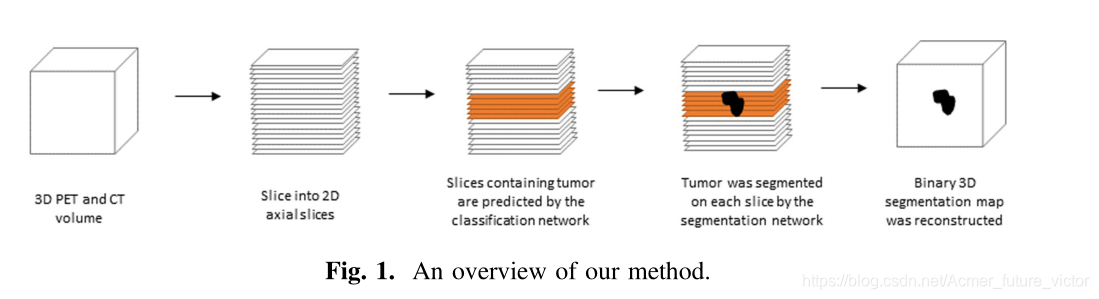
Classification Network
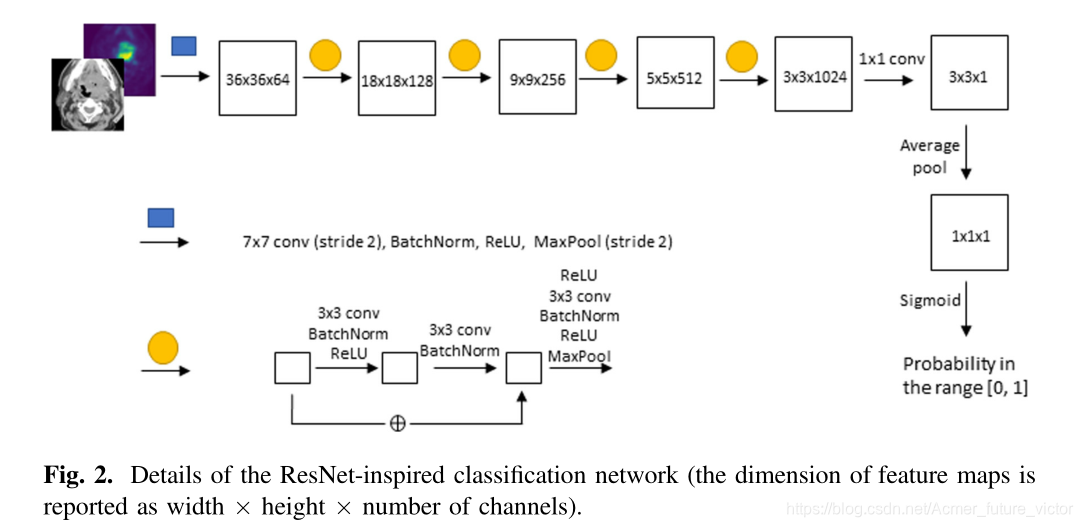
分类网络的目标是选择可能包含GTVT的轴向切片,使得分割网络可以集中在分割上。这种受ResNet启发的网络体系结构如图2所示:大小为(144,144,2)的输入切片首先经历7x7Conv-BatchNorm-RELU-MaxPool块,然后是具有剩余单元的一系列3x3Conv-BatchNorm-RELU块[4]。最后,3x3x1特征图经过平均池化和sigmoid运算,以产生范围在[0.0,1.0]之间的数字,对应于切片包含GTVt的可能性。用训练集中的所有轴向切片训练分类网络。在训练过程中,以总体精度的二进制交叉熵作为损失函数。关于培训的详细信息见4.1节。
Segmentation Network
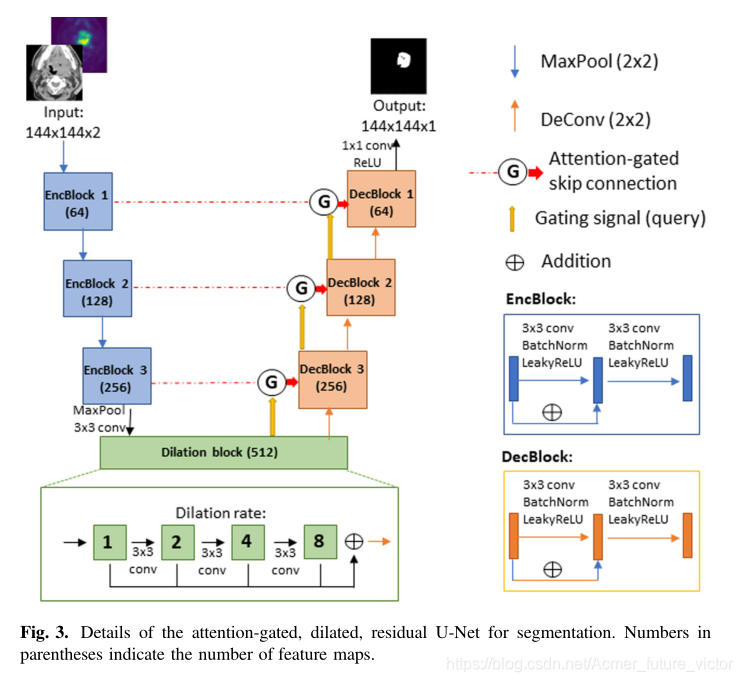
分割网络的目标是在给定包含GTVt的可能性很高的2D切片的情况下输出分割图。如图3所示,该网络结构类似于U-Net,它使用带有跳跃连接的编码器-解码器来促进反向传播期间改进的梯度流[5]。此外,注意门控、扩展卷积和残差单元等其他特征也被纳入其中,因为在先前的研究中,它们已经被证明可以改善U-net的分割性能。Oktay et al.结果表明,在U-net中加入注意力门控显著改善了CT扫描上胰腺分割的性能[6],这一特征可能有助于我们的研究,使该模型能够基于CT特征和PET亲和力有效地识别肿瘤。扩张卷积已被证明在扩大网络的接受范围方面很有用[7],这对本研究的任务很重要,因为网络需要学会忽略不能发生头颈部癌症的部位(例如大脑)的PET亲和力。此外,除了跳跃连接外,残差单元还用于在反向传播过程中允许额外的梯度流[4]。
分割网络仅用包含GTVt的切片进行训练。训练过程中采用soft Dice Loss作为损失函数。关于培训的详细信息见4.1节。
Experiment
Training Details
将201例患者随机分为181例进行训练,20例进行验证,共有26004层CT和PET轴位图像可供训练,其中6379层包含GTVt.
分类网络在包含GTVt的切片(“正切片”)和不包含GTVt的切片(“负切片”)上进行训练。然而,由于两个类之间的数据不平衡,我们在训练过程中对负片进行了欠采样;因此,每个epoch包含全部6,379个“正片”和6,379个随机抽样的“负片”。批大小为10。使用ADAM作为优化器,学习率为0.005,β1为0.9,β2为0.999,epsilon为1e-7。当验证集的总体分类精度连续2个epoch下降时,停止训练以减少过拟合。在推理过程中,采用0.5的阈值进行分类。
分割网络仅使用“正切片”进行训练。使用ADAM作为优化器,学习率为0.005,β1为0.9,β2为0.999,epsilon为1e-7,batchsize为1。网络训练了20个epoch,并使用验证集上Dice相似系数最高的epoch的模型进行推理,以避免过拟合。在推理过程中,所有像素的阈值均为0.5用于分割。
该程序是以TensorFlow2.2为框架在Python3.8中实现的。网络培训在Google Colab和NVIDIA Tesla V100 GPU上进行。
Results
在验证数据集上独立地选择和组合性能最好的分类和分割模型来构建最终模型。以整个3D体积作为模型输入,最终的模型在由53名患者组成的测试集中实现了0.644的平均DSC、0.694的平均精度和0.667的平均召回率。
我们描述了一种基于两阶段卷积神经网络的头颈部原发肿瘤大体肿瘤体积自动分割方法。输入图像首先通过分类网络来预测肿瘤的存在,如果是,则通过第二分割网络来产生二进制分割图。利用这种方法,我们获得了很好的Dice相似系数。