小罗碎碎念
这篇文章介绍了一种利用深度学习技术进行虚拟组织染色的方法,能够快速、高效地将未标记的尸检组织切片转化为数字化的H&E染色图像,提高病理分析的质量和效率。

| 作者角色 | 姓名 | 单位名称 |
|---|---|---|
| 第一作者 | Yuzhu Li | 加利福尼亚大学洛杉矶分校(University of California, Los Angeles) |
| 第一作者 | Nir Pillar | 加利福尼亚大学洛杉矶分校(University of California, Los Angeles) |
| 第一作者 | Jingxi Li | 加利福尼亚大学洛杉矶分校(University of California, Los Angeles) |
| 通讯作者 | Aydogan Ozcan | 加利福尼亚大学洛杉矶分校(University of California, Los Angeles) |
文章详细描述了尸检过程的重要性,以及传统染色方法面临的挑战,包括染色伪影、成本高、耗时长和劳动密集。
研究者们通过训练一个神经网络模型,将未标记的组织切片的自发荧光图像转化为数字化的H&E染色图像,有效避免了自溶引起的染色伪影。
此外,文章还介绍了一种名为RegiStain的数据高效深度学习框架,它结合了图像配准神经网络和虚拟染色网络的并行训练,以提高图像配准的准确性。
研究结果表明,即使在训练时仅使用保存良好的组织样本,该虚拟染色网络也能快速准确地对经历严重自溶的无标记肺组织切片进行虚拟H&E染色,包括COVID-19引起的肺炎样本。通过与保存良好的组织区域的H&E组织化学染色图像进行比较,定量评估虚拟染色图像的质量,结果显示虚拟染色结果与传统染色结果之间没有统计学上的显著差异。
文章还讨论了虚拟染色技术的潜在应用,包括在全球健康危机期间快速准确的尸检样本分析,以及对坏死组织的染色,这些组织在组织化学染色中面临类似的挑战。总的来说,这项技术为病理学研究和临床诊断提供了一种快速、资源高效的解决方案,有望在未来的组织样本分析中发挥重要作用。
一、利用深度学习对未标记尸检组织切片进行虚拟H&E染色
常规的苏木精和伊红(H&E)组织化学染色(顶部)需要由病理技术员执行化学样本处理程序,这些程序耗时、劳动密集、成本高昂,并且在尸检样本组织自溶的情况下容易造成染色失败。
相比之下,基于深度学习的虚拟染色神经网络(底部)可以用来对未标记尸检组织切片进行快速、成本效益高且准确的虚拟染色,基于它们的自发荧光显微镜图像,即使在组织化学染色失败的 自溶组织区域也能提供高质量的染色结果。
1-1:传统方式VS虚拟染色方式
这张图展示了两种不同的组织切片染色方法:传统的苏木精-伊红(H&E)染色和基于深度学习的虚拟染色技术。

传统H&E染色流程:
- 组织标本:尸检组织样本。
- 固定:使用福尔马林处理组织,以保持其结构。
- 切片:将固定后的组织切成薄片。
- H&E染色:使用苏木精和伊红进行染色。苏木精主要染色细胞核,伊红主要染色细胞质和其他组织结构。
- 组织切片:染色后的组织切片。
- 显微成像(明场):使用显微镜观察并拍摄染色后的组织切片。
- H&E染色组织图像:最终得到的H&E染色图像,用于病理学分析。
深度学习虚拟染色流程:
- 未染色的组织切片:与H&E染色流程中的切片相同,但没有进行染色。
- 显微成像(自发荧光):使用显微镜观察未染色组织切片的自发荧光。
- 自发荧光组织图像:记录自发荧光的图像。
- 虚拟染色网络:利用深度学习算法,将自发荧光图像转换为类似H&E染色的图像。
- 虚拟H&E组织图像:最终得到的虚拟染色图像,模拟了H&E染色的效果。
技术对比:
- 传统H&E染色:需要使用化学染料,步骤繁琐,且染色过程可能对环境和操作人员有潜在危害。
- 虚拟染色:通过深度学习技术,可以减少化学染料的使用,简化染色过程,同时可能提高染色的一致性和可重复性。
这种虚拟染色技术的发展,为病理学研究和诊断提供了新的工具,尤其是在资源有限或需要快速诊断的情况下。
1-2:训练+验证
在作者采用的训练策略中,虚拟染色网络和图像配准网络的训练是相互优化的;这种训练仅使用保存良好的尸检样本(在COVID-19之前收集的)进行,一旦训练完成,虚拟染色网络模型就被用来成功染色来自COVID-19以及非COVID-19尸体的严重自溶的尸检样本。

训练阶段:
- 输入(无标签):使用DAPI和TxRed染色的组织样本图像作为输入。
- 虚拟染色网络(G):深度学习模型,用于将输入图像转换为类似H&E染色的图像。
- 配准分析网络(R):用于对输入图像和真实H&E染色图像进行配准,以确保两者在空间上的一致性。
- 真实H&E图像:作为训练的“真实”或“地面真实”数据,用于指导和评估虚拟染色网络的性能。
- RegiStain训练框架:结合虚拟染色网络和配准分析网络的对称训练框架,用于优化模型性能。
测试阶段:
-
COVID-19肺炎(严重肺泡损伤):
- 输入(无标签):使用DAPI和TxRed染色的COVID-19肺炎组织样本图像。
- 虚拟染色网络:应用训练好的模型将输入图像转换为类似H&E染色的图像。
- 输出:生成的虚拟H&E染色图像,用于分析和诊断。
-
非COVID-19肺炎(严重肺泡损伤):
- 输入(无标签):使用DAPI和TxRed染色的非COVID-19肺炎组织样本图像。
- 虚拟染色网络:同样应用训练好的模型进行转换。
- 输出:生成的虚拟H&E染色图像,用于分析和诊断。
技术细节:
- DAPI:一种荧光染料,常用于染色细胞核。
- TxRed:一种荧光染料,用于染色细胞质或其他组织结构。
- 虚拟染色网络:通过深度学习技术,将无标签的组织图像转换为类似H&E染色的图像,这种方法可以减少对化学染料的依赖,提高染色过程的效率和可重复性。
- 配准分析网络:确保虚拟染色图像与真实H&E图像在空间上的一致性,这对于准确的病理分析至关重要。
这种方法的应用可以提高病理诊断的效率,尤其是在资源有限或需要快速诊断的情况下。同时,它也为研究提供了一种新的工具,可以用于分析和比较不同疾病状态下的组织变化。
二、RegiStain框架用于训练尸检虚拟染色模型
这张图展示了一个基于生成对抗网络(GAN)的虚拟染色和配准分析框架,用于将无标签的组织图像(DAPI和TxRed染色)转换为类似H&E染色的图像,并且进行精细的空间配准。
在训练过程中,生成对抗网络(GAN)模块,包括虚拟染色网络(G)和判别器网络(D),与图像配准分析网络(R)一起训练,以确保从输入的自发荧光组织图像到H&E等效明场图像的像素级精确学习。
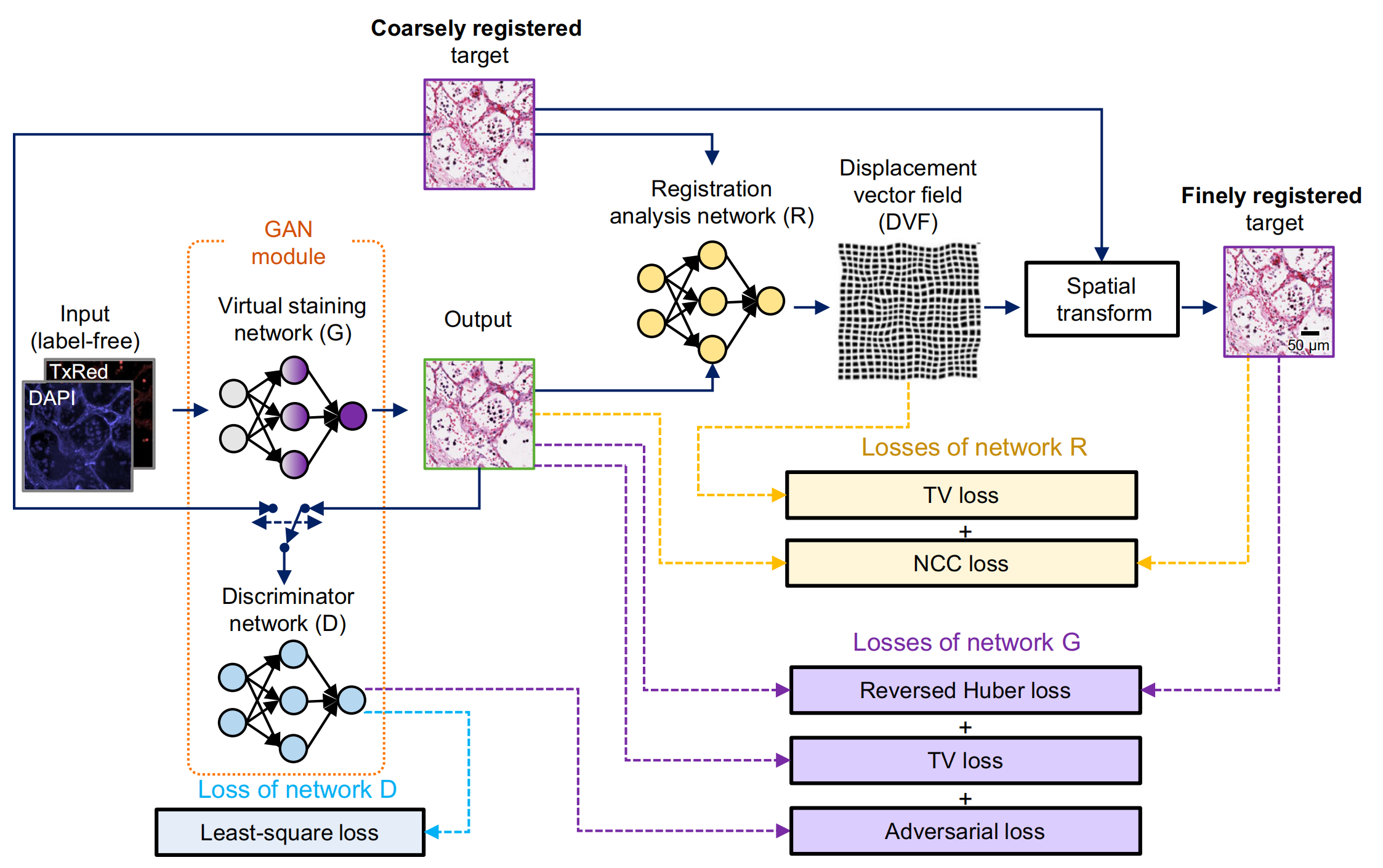
以下是对图中各部分的详细分析:
2-1:GAN模块
- 输入(无标签):DAPI和TxRed染色的组织图像作为输入。
- 虚拟染色网络(G):生成对抗网络的生成器部分,负责将输入图像转换为类似H&E染色的图像。
- 输出:经过虚拟染色网络处理后的图像。
- 判别器网络(D):生成对抗网络的判别器部分,用于区分生成的图像和真实H&E染色图像。
2-2:配准分析网络(R)
- 粗略配准目标:初始的H&E染色图像,用于与生成的图像进行比较。
- 配准分析网络(R):负责计算输入图像与目标图像之间的位移向量场(DVF),以实现空间配准。
- 位移向量场(DVF):描述了输入图像中每个点到目标图像中对应点的位移。
- 空间变换:应用位移向量场对输入图像进行空间变换,以实现精细配准。
2-3:损失函数
-
网络R的损失:
- TV损失:总变差损失,用于保持图像的平滑性。
- NCC损失:归一化互相关损失,用于衡量两个图像之间的相似度。
-
网络G的损失:
- 反向Huber损失:一种鲁棒的损失函数,用于处理异常值。
- TV损失:与网络R中的TV损失相同,用于保持生成图像的平滑性。
- 对抗损失:用于训练生成器生成更逼真的图像,以欺骗判别器。
-
网络D的损失:
- 最小二乘损失:用于训练判别器,使其能够更准确地区分真实图像和生成图像。
2-4:流程
- 输入图像首先通过虚拟染色网络G进行处理,生成类似H&E染色的图像。
- 生成的图像与粗略配准目标进行比较,通过配准分析网络R计算位移向量场。
- 位移向量场用于对生成的图像进行空间变换,实现精细配准。
- 整个过程中,通过不同的损失函数来优化网络G和R的性能,确保生成的图像既逼真又与目标图像精确对齐。
这个框架结合了深度学习和图像处理技术,用于提高组织图像分析的准确性和效率,特别是在病理学研究和诊断中。
三、未标记尸检组织切片的虚拟H&E染色图像与相应组织化学H&E染色结果之间的视觉比较
【DAPI和TxRed】+【虚拟HE】+【真实HE】

三个示例局部区域的放大图像

注意:
- d-f是使用DAPI通道捕获的这些区域的自发荧光图像,g-i是使用TxRed通道捕获的相应图像。这些DAPI和TxRed自发荧光图像对作为我们虚拟染色网络的输入。
- j-l是与d-f(或g-i)相同区域的虚拟H&E染色结果,这些结果是基于d和g、e和h、以及f和i,分别由模型的虚拟染色网络数字生成的。
- m-o是与j-l相同区域的组织化学H&E染色结果,它们在细胞核中显示出染色不足的伪迹。
- 不同类型的细胞,包括中性粒细胞、淋巴细胞和巨噬细胞的染色结果,在图像中使用不同方向的箭头进行标注。
- 本图中产生这些代表性图像的过程重复进行,盲测试阶段所有10张尸检切片(n = 10)的结果相似。
四、病理学家进行的基于评分的定量评估
四位获得董事会认证的病理学家从不同方面评估的虚拟和组织化学染色的苏木精和伊红(H&E)图像的染色质量评分。

这些方面包括染色伪迹、细胞外细节、细胞质细节和细胞核细节。评估的染色质量评分范围从1到4,其中4代表完美,3代表非常好,2代表可接受,1代表不可接受。这些评分的均值和标准差值是在所有100个测试样本视场(FOVs)和四位病理学家(n = 100 × 4 = 400)之间计算的。
小提琴图显示了使用所有100个测试FOVs评估的细胞密度评分的分布。

评估的细胞密度评分范围从1到4,其中4代表显著高的细胞计数,3代表大量的细胞,2代表适量的细胞,1代表低的细胞计数。